Во время эмбриогенеза эпигенетические часы показывают событие омоложения с последующим старением
Авторы: Csaba Kerepesi, Bohan Zhang, Sang-Goo Lee, Alexandre Trapp, Vadim N. Gladyshev
Введение
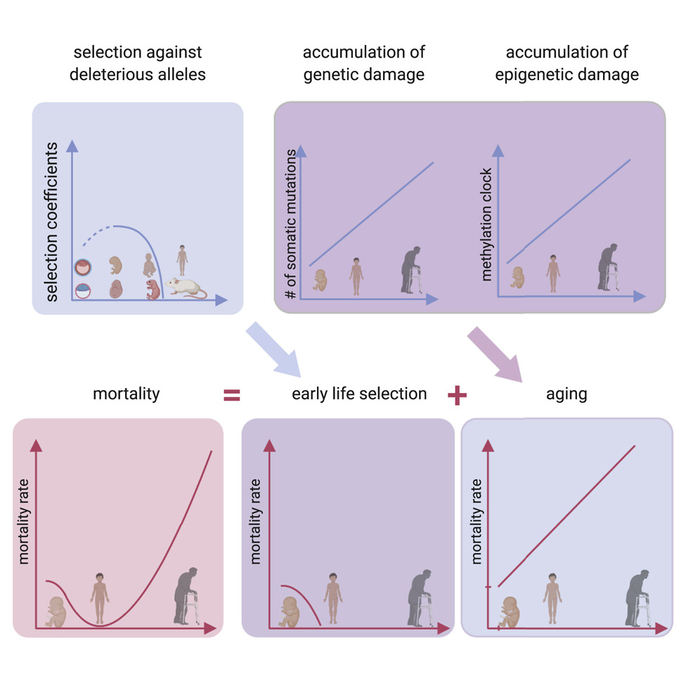
Старение характеризуется прогрессирующим накоплением повреждений, что приводит к потере физиологической целостности, нарушению функции и повышенной уязвимости к смерти (1). Хотя процесс старения влияет на весь организм, часто обсуждается, что зародышевая линия не стареет, потому что эта линия бессмертна в том смысле, что зародышевая линия воспроизводится бесконечно с начала жизни (2–4). Это понятие восходит к 19 веку, когда Август Вейсманн предложил разделить нестареющую зародышевую линию и стареющую сому. Однако, пребывая в метаболически активном состоянии в течение двух или более десятилетий, прежде чем внести свой вклад в потомство, зародышевые линии человека накапливают молекулярные повреждения, такие как модифицированные долгоживущие белки, эпимутации, побочные продукты метаболизма и другие вредные изменения, связанные с возрастом (5 , 6). Было показано, что сперматозоиды демонстрируют отчетливую картину возрастных изменений (7–9). Соответственно, недавно было высказано предположение, что клетки зародышевой линии могут стареть и омолаживаться у потомства после зачатия (10,11). Если это так, должна быть точка (или период) самого низкого биологического возраста (здесь называется нулевой точкой) во время начальных фаз эмбриогенеза (рис. 1А). Здесь мы провели количественную оценку данных для проверки этой идеи.
Благодаря последним достижениям в области технологий машинное обучение процветает и привело к прорывам во многих областях науки, обнаружив многомерные взаимосвязи (12). Области старения и биологии развития также использовали потенциал машинного обучения, разрабатывая алгоритмы («часы старения»), которые могут оценивать хронологический или биологический возраст (то есть возраст на основе молекулярных маркеров) организма по заданным данным (13, 14). . Поскольку эпигеномные изменения, которые приводят к нарушению регуляции транскрипционных и хроматиновых сетей, являются ключевыми компонентами старения (15), эпигенетические часы, основанные на уровнях метилирования определенных сайтов CpG, стали многообещающим молекулярным средством оценки биологического возраста (16, 17). Было показано, что эти часы позволяют количественно измерять многочисленные аспекты старения человека (17–22). Например, эпигенетическое ускорение возраста было связано с возрастными состояниями, такими как смертность от всех причин (23, 24), когнитивная производительность (25), слабость (26), болезнь Паркинсона (27), синдром Дауна (28) и Синдром Вернера (29). Часы эпигенетического старения также были разработаны для мышей и могут использоваться для оценки вмешательств, направленных на увеличение продолжительности жизни, таких как ограничение калорий и нокаут рецепторов гормона роста (30–36). Наиболее важно то, что часы, разработанные на основе паттернов старения в основном взрослых тканей, сообщают об эффектах клеточного омоложения при полном или частичном перепрограммировании взрослых фибробластов в индуцированные плюрипотентные стволовые клетки (ИПСК), что продемонстрировано как на людях, так и на мышах (33, 35, 37). –39). Несмотря на то, что ИПСК соответствуют эмбриональному состоянию и переход к этим клеткам включает в себя серьезные молекулярные изменения, включая изменения в эпигеноме, это событие омоложения можно оценить с помощью эпигенетических часов. Совсем недавно универсальные часы для млекопитающих были разработаны на основе консервативных цитозинов, уровни метилирования которых изменяются с возрастом у всех видов млекопитающих (40).
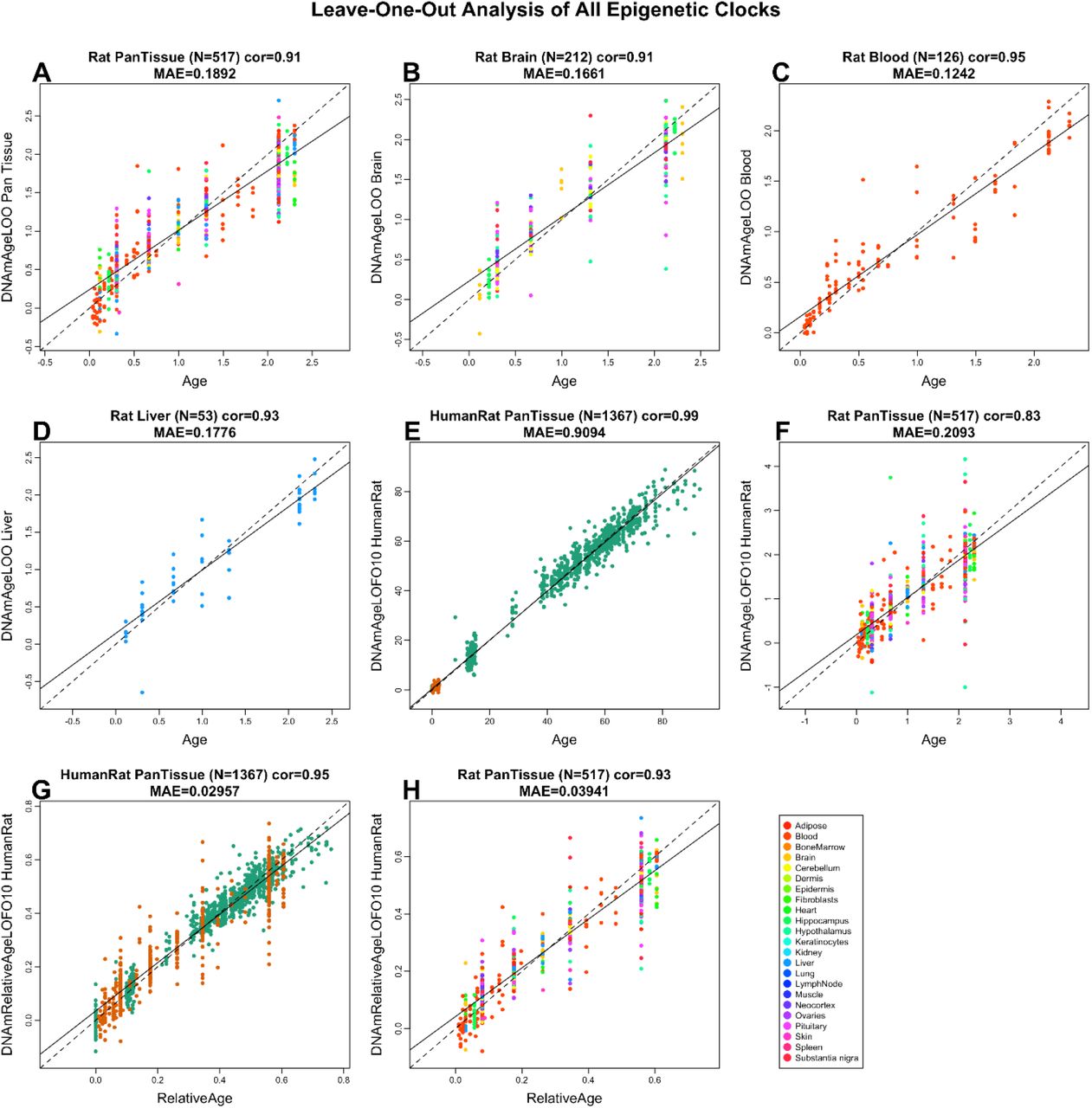
Учитывая, что эпигенетические часы отслеживают процесс старения, их можно применять к раннему развитию, чтобы характеризовать динамику биологического возраста в течение этого периода жизни. Недавние исследования показали, что часы могут быть успешно применены к развитию плода человека с использованием образцов головного мозга, сетчатки и пуповинной крови (41–43). Однако эпигенетическая возрастная динамика на протяжении всего пренатального развития для всего организма оставалась неизученной. Здесь мы разработали новые эпигенетические часы для нескольких тканей с использованием машинного обучения и применили их вместе с другими существующими часами старения для оценки пренатального развития млекопитающих с точки зрения старения. Этот подход выявил период омоложения во время раннего эмбриогенеза и время начала старения у млекопитающих.
ПОЛУЧЕННЫЕ РЕЗУЛЬТАТЫ
Временное перепрограммирование восстанавливает молодую экспрессию генов в старых адипогенных клетках
Чтобы оценить эффекты временного перепрограммирования с использованием полного набора факторов Яманака, мы разработали полицистронный тетрациклин-индуцибельный лентивирусный вектор SOKM с конститутивным флуоресцентным репортером (LTV-Y4TF, рис. 1A) [19]. Мы использовали LTV-Y4TF и лентивирус-трансактиватор тетрациклина (LTV-Tet) для трансдукции первичных адипогенных клеток от молодых (2-4 месяца) и старых (20-24 месяцев) мышей C57Bl / 6 in vitro. После трансдукции мы выполнили пульс-чейз, добавляя доксициклин (Dox) в среду для культивирования клеток в течение 3 дней и набдюдая в течение 3 дней (рис. 1B). Мы использовали множественность заражения (multiplicity of infection, MOI), достаточную для трансдукции 50% клеток в каждом состоянии, так что часть клеток в каждой лунке подвергается воздействию Dox, но не содержит обоих трансгенов, необходимых для перепрограммирования. Таким образом, эти клетки служат контролем in situ. Эта экспериментальная схема обеспечивает более короткий индукционный период и более длительный период отслеживания, чем в предыдущих сообщениях [9, 10, 11]. После периода отслеживания мы отсортировали двойные трансген-положительные (Tg +) и одиночные трансген-отрицательные (Tg-) клетки каждого возраста с помощью цитометрии и профилировали клеточные транскриптомы с помощью отдельноклеточного RNA-seq (методы).
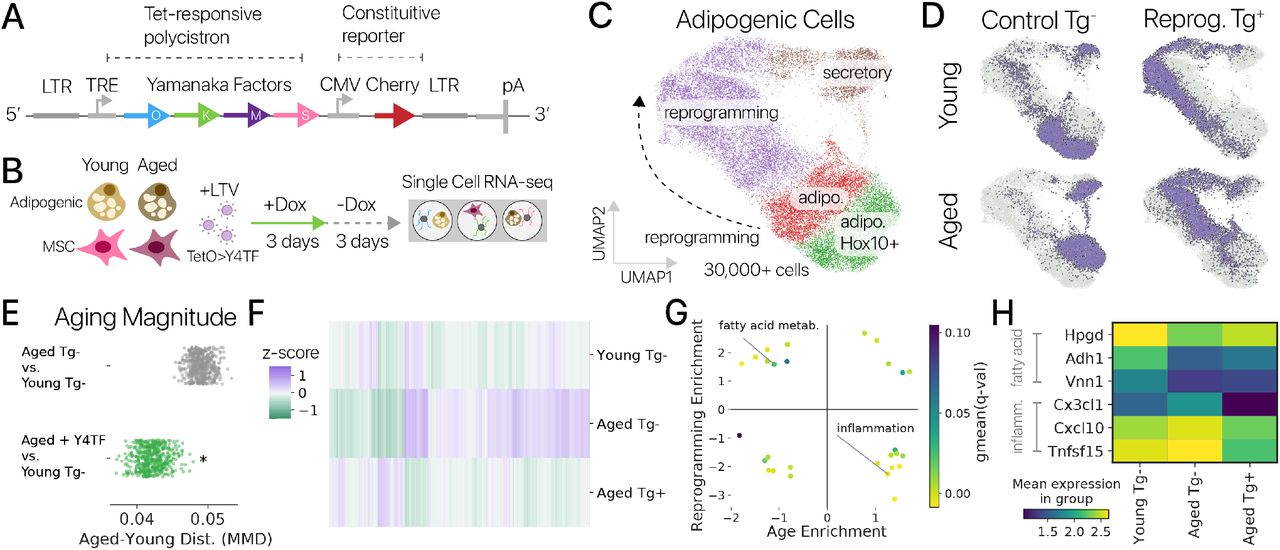 Рисунок 1: Временное плюрипотентное перепрограммирование восстанавливает молодую экспрессию генов в адипогенных клетках мышей. (A) Схематическая диаграмма нашего плюрипотентного перепрограммирующего лентивирусного вектора (LTV-Y4TF). Экспрессия полицистрона фактора Яманака контролируется элементом ответа на тетрациклин (TRE). (B) Схема нашего эксперимента по временному плюрипотентному перепрограммированию. Мы провели 3-дневный импульс / 3-дневную погоню за SOKM с использованием индуктора Dox в адипогенных клетках и мышечных МСК молодых и старых мышей. После погони клетки были профилированы с помощью отдельноклеточного РНК-секвенирования. (C) UMAP-проекция профилей мРНК отдельных клеток из молодых и старых адипогенных клеток. Состояния выражений, специфичные для управления и перепрограммирования, аннотируются. Адипогенные состояния маркируются Lpl, секреторные состояния - Rspo1, а специфические состояния перепрограммирования - Snca и Nanog. (D) Контрольная (Tg-) и перепрограммированная (Tg +) популяции молодых и старых клеток выделены во вложении. Контрольные адипогенные клетки делятся на подмножества Hoxc10 +/-. Легко различить молодые и пожилые популяции. (E) Величина связанных с возрастом изменений в экспрессии генов была значительно снижена при обработке Y4TF, как измерено с использованием максимального среднего расхождения (MMD, p 0,001, сумма рангов Вилкоксона). Поэтому старые перепрограммированные клетки ближе к молодому контрольному состоянию, чем старые контрольные клетки. (F) Временное перепрограммирование (Aged Tg +) восстанавливает молодой паттерн экспрессии генов по тысячам генов (значительное изменение в направлении молодости, q 0,10). (G) Анализ обогащения набора генов показывает, что многие генные программы, выключающиеся с возрастом, активируются посредством перепрограммирования и наоборот. Метаболизм жирных кислот был одной из программ молодости , восстанавливаемых перепрограммированием, а воспалительные реакции, усиленные с возрастом, подавлялись перепрограммированием (наборы генов MSigDB Hallmark). (H) Гены программ метаболизма жирных кислот и воспалительной реакции показывают смягчение возрастных изменений во временно перепрограммированных клетках.
Рисунок 1: Временное плюрипотентное перепрограммирование восстанавливает молодую экспрессию генов в адипогенных клетках мышей. (A) Схематическая диаграмма нашего плюрипотентного перепрограммирующего лентивирусного вектора (LTV-Y4TF). Экспрессия полицистрона фактора Яманака контролируется элементом ответа на тетрациклин (TRE). (B) Схема нашего эксперимента по временному плюрипотентному перепрограммированию. Мы провели 3-дневный импульс / 3-дневную погоню за SOKM с использованием индуктора Dox в адипогенных клетках и мышечных МСК молодых и старых мышей. После погони клетки были профилированы с помощью отдельноклеточного РНК-секвенирования. (C) UMAP-проекция профилей мРНК отдельных клеток из молодых и старых адипогенных клеток. Состояния выражений, специфичные для управления и перепрограммирования, аннотируются. Адипогенные состояния маркируются Lpl, секреторные состояния - Rspo1, а специфические состояния перепрограммирования - Snca и Nanog. (D) Контрольная (Tg-) и перепрограммированная (Tg +) популяции молодых и старых клеток выделены во вложении. Контрольные адипогенные клетки делятся на подмножества Hoxc10 +/-. Легко различить молодые и пожилые популяции. (E) Величина связанных с возрастом изменений в экспрессии генов была значительно снижена при обработке Y4TF, как измерено с использованием максимального среднего расхождения (MMD, p 0,001, сумма рангов Вилкоксона). Поэтому старые перепрограммированные клетки ближе к молодому контрольному состоянию, чем старые контрольные клетки. (F) Временное перепрограммирование (Aged Tg +) восстанавливает молодой паттерн экспрессии генов по тысячам генов (значительное изменение в направлении молодости, q 0,10). (G) Анализ обогащения набора генов показывает, что многие генные программы, выключающиеся с возрастом, активируются посредством перепрограммирования и наоборот. Метаболизм жирных кислот был одной из программ молодости , восстанавливаемых перепрограммированием, а воспалительные реакции, усиленные с возрастом, подавлялись перепрограммированием (наборы генов MSigDB Hallmark). (H) Гены программ метаболизма жирных кислот и воспалительной реакции показывают смягчение возрастных изменений во временно перепрограммированных клетках.
После фильтрации по качеству мы получили в распоряжение 30 000+ профилей мРНК адипогенных клеток. После шумоподавления и уменьшения размерности с помощью вариационного автоэнкодера (методы) [20] мы сгруппировали профили клеток и обнаружили, что перепрограммирование индуцирует набор новых состояний экспрессии генов, невидимых в контрольных клетках (рис. 1C, D). Контрольные клетки находились в адипогенном состоянии, обозначенном Lpl, и секреторном состоянии, обозначенном Rspo1. Подавляющее большинство обработанных клеток занимали клеточные состояния, специфичные для перепрограммирования, что позволяет предположить, что эффекты временного перепрограммирования обладают высокой проникающей способностью. Молодые и старые клетки занимали отдельные области латентного пространства как в контрольной, так и в перепрограммированной популяциях, аналогично предыдущему исследованию контрольных клеток in vivo, предполагая, что некоторые черты старения сохраняются после перепрограммирования (рис. 1D; рис. S1) [21]. . Различия между животными были небольшим источником вариаций (1%, рис. S2, ANOVA, методы).
Сначала мы хотели определить, уменьшилось ли транскрипционное расстояние между молодыми и старыми контрольными клетками - степень старения - при временном перепрограммировании. Мы измерили степень старения, используя максимальное среднее несоответствие (MMD), статистику репрезентативного обучения, которая измеряет сходство двух популяций и обеспечивает тест на статистическую значимость [22, 23]. Здесь мы вычислили MMD по популяциям клеток, используя скрытые переменные автоэнкодера, чтобы зафиксировать изменения во всем транскриптоме. Мы сравнили как старые контрольные, так и старые перепрограммированные клетки с молодыми контрольными клетками с помощью MMD и обнаружили, что степень старения значительно снизилась после проведения перепрограммирования (рис. 1E, p 0,001, сумма рангов Вилкоксона; методы). Это уменьшение различия молодых и старых клеток демонстрирует, что временное перепрограммирование может восстановить молодой рисунок экспрессии генов в транскриптоме.
Чтобы определить, какие гены вызывают это изменение, мы выполнили анализ дифференциальной экспрессии и обнаружили, что перепрограммирование вызывало значительные изменения уровня экспрессии в направлении молодости в 3485 генах из 5984 генов, измененных с возрастом (рис. 1F). Перепрограммирование вызывало омоложение значительно чаще, чем можно было ожидать в случае случайных изменений (биномиальный тест, p 0,0001). Мы использовали анализ обогащения набора генов [24] для сравнения транскрипционных эффектов старения и перепрограммирования и обнаружили, что перепрограммирование противодействует возрастным изменениям во многих наборах генов (рис. 1G). Двумя наиболее яркими примерами этого феномена были набор генов адипогенного «метаболизма жирных кислот», подавляющийся с возрастом и активируемый перепрограммированием, и набор генов «воспалительного ответа», активированный с возрастом и подавляющийся перепрограммированием (рис. 1H). Временное перепрограммирование может реконструировать эпигеном, чтобы позволить реактивацию подавленной программы жирных кислот. Мы обнаружили аналогичное подавление адипогенных генов с возрастом в данных по отдельным клеткам, собранным in vivo (рис. S1) [21]. Восстановление этой характерной метаболической программы предполагает, что некоторые функции старых адипогенных клеток могут быть улучшены временным перепрограммированием.
Идентичность клетки диктует эффекты временного перепрограммирования
Затем мы задались вопросом, было ли восстановление молодой экспрессии генов постоянной особенностью временного перепрограммирования в случае разных типов клеток. Мы провели эксперименты по временному перепрограммированию мезенхимальных стволовых клеток мышечного происхождения [25] для исследования этого вопроса. Мы получили более 20000 профилей МСК и обнаружили, что молодые и старые контрольные клетки были менее различимы, чем в случае адипогенных клеток (рис. S3A, B), но возраст клеток все еще можно было легко определить с помощью классификационной модели (точность 93%, рис. S4). , Методы). Подобно нашим экспериментам на адипогенных клетках, временное перепрограммирование индуцировало новый набор состояний экспрессии генов, специфичных для перепрограммирования, и отсутствующих в контрольных клетках обоих возрастов (рис. S3C). Эти состояния характеризовались подавлением программы перехода эпителия в мезенхиму (EMT) (обогащение набора генов Hallmark из 150 основных дифференциальных генов; точный тест Фишера; q 0,0001). Мы также обнаружили, что молодая экспрессия генов была восстановлена в 712 генах (значительное изменение в направлении молодости), программа фиброзных генов, вызванная старением, была подавлена, а количественная оценка уровня старения, полученная на основе данных массивных последовательностей РНК, уменьшилась (рис. . S5).
Однако, когда мы вычислили величину старения в MSC, мы обнаружили, что старые клетки были более отличными от молодых контрольных после перепрограммирования (Рис. S3F). Этот результат, вероятно, был связан с тем, что более 4000 генов были значительно изменены при перепрограммировании, но не с возрастом. Мы предположили, что в MSC степень старения может быть меньше, чем в адипогенных клетках, так что ортогональные эффекты перепрограммирования доминируют в контексте MSC. Мы проверили эту гипотезу, сравнив величину старения в контрольных адипогенных клетках и MSC в общем латентном транскрипционном пространстве и обнаружили, что старение вызывает значительно больший сдвиг экспрессии генов в адипогенных клетках (рис. S6A, C; методы). Эти результаты предполагают, что в некоторых типах клеток временные эффекты перепрограммирования, не связанные с возрастом клеток, могут доминировать.
Мы также предположили, что перепрограммирование может вызывать эффекты, специфичные для некоторых типов клеток. Чтобы проверить эту гипотезу, мы выполнили анализ дифференциальной экспрессии для выявления как перепрограммирование влияет на разные типы клеток (методы). Этот анализ выявил более 10 000 генов, для которых перепрограммирование вызывало существенно разные эффекты (q 0,05, тест отношения правдоподобия, регрессия препятствий). Мы также обнаружили, что 11% неостаточных вариаций объясняются специфическими для типов клеток эффектами перепрограммирования в общем латентном транскрипционном пространстве (рис. S6B, ANOVA).
Чтобы выяснить влияние типа клеток на результаты временного перепрограммирования, мы извлекли главные гены со значимым разбросом влияния перепрограммирования на разные типы клеток и выполнили анализ онтологии генов. Программы внеклеточного матрикса и EMT были подавлены перепрограммированием в обоих типах клеток, но значительно меньше в адипогенных клетках. Напротив, наборы генов гипоксии и гликолиза в большей степени регулируются перепрограммированием в MSC, чем в адипогенных клетках (рис. S6E, q 0,05, точный тест Фишера). Исследование отдельных генов подтвердило влияние типа клеток на эффекты перепрограммирования в этих генных программах (рис. S6D, F). Таким образом, идентичность соматических клеток определяет эффекты временного перепрограммирования многих генов, предполагая, что молодой паттерн экспрессии генов в одних типах клеток может быть восстановлен более эффективно, чем в других.
Идентичности соматических клеток подавляются временным перепрограммированием
Основываясь на наших наблюдениях за новыми состояниями клеток во временно перепрограммированных адипогенных клетках и MSC, мы предположили, что регуляторные сети генов, обеспечивающие идентичность соматических клеток, могут быть репрессированы, даже если факторы перепрограммирования экспрессируются только временно. Учитывая континуум состояний перепрограммирования, которые мы наблюдали, непрерывная система координат, назначающая каждой ячейке позицию на траектории перепрограммирования, позволяет проводить скупой анализ. Чтобы построить систему координат для кратковременного перепрограммирования, мы использовали псевдовременной анализ, метод для вывода непрерывных процессов с использованием отношений между ячейками, профилированных в один момент времени (рис. 2A, C; методы) [26].
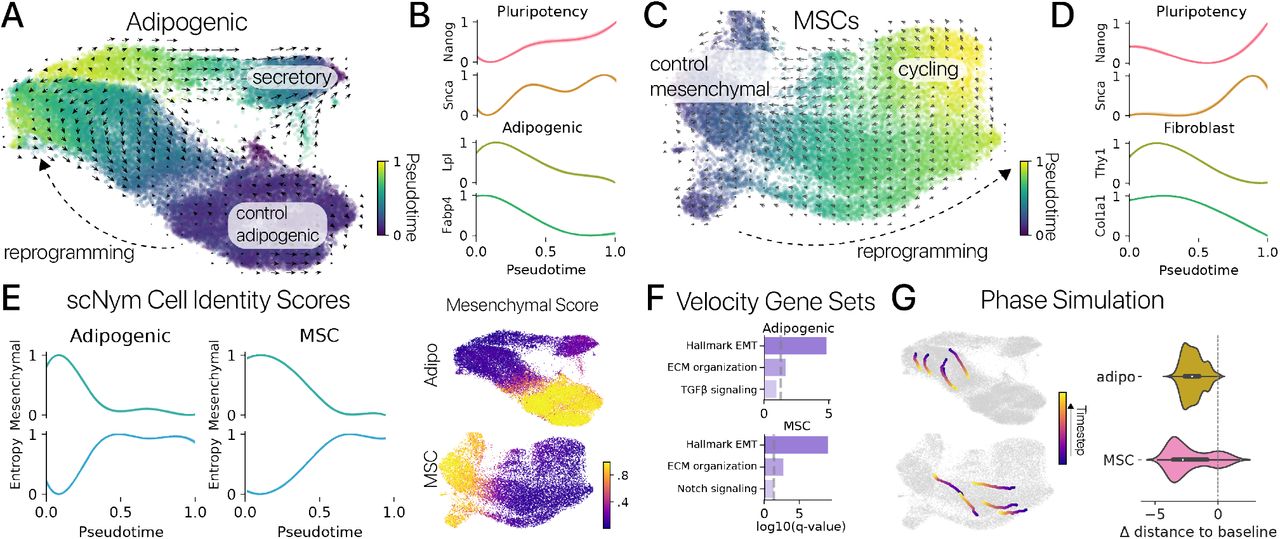 Рисунок 2: Идентичность соматических клеток подавляется временным перепрограммированием и повторно приобретается посредством вторичной дифференцировки.(A) Временное перепрограммирование в адипогенных клетках индуцирует непрерывную траекторию перепрограммирования специфичных клеточных состояний. Мы зафиксировали эту траекторию количественно с помощью диффузионной псевдовременной координаты. Скорость РНК (стрелки) предсказывала, что клетки в специфичных для перепрограммирования состояниях дифференцируются в направлении идентичности контрольных клеток. (B) Связанные с плюрипотентностью гены Nanog и Snca были индуцированы в наиболее дистальных состояниях перепрограммирования, в то время как гены идентичности адипоцитов Lpl и Fabp4 были подавлены (значения соответствия GAM, среднее ± 95% ДИ). (C) MSC, полученные из мышц, демонстрируют сходную траекторию состояний перепрограммирования. Скорость РНК аналогичным образом предсказывала, что MSC в специфичных для перепрограммирования состояниях повторно дифференцируются в направлении своей исходной идентичности соматических клеток. (D) Гены, ассоциированные с плюрипотентностью, также индуцировались в дистальных состояниях перепрограммирования MSC, в то время как гены мезенхимальной идентичности Thy1 и Col1a1 подавлены. (E) Программная активность мезенхимальной идентичности, выведенная с использованием классификаторов типов клеток и набора данных атласа клеток мыши, оцененных по псевдовремени (слева) и проецируемых в латентные пространства транскрипции (справа). Программы мезенхимальной идентичности подавлялись в дистальных состояниях перепрограммирования для обоих типов клеток, в то время как энтропия идентичности клеток возрастала (p 0,01, тест Вальда). (F) Обогащенный анализ онтологии генов показывает, что эпителиально-мезенхимальный переход (EMT), внеклеточный матрикс (ECM) и гены передачи сигналов клетки являются основными движущими силами скорости РНК. (G) Траектории состояния клеток моделировались на основе оценок скорости РНК в каждом типе клеток. Смоделированные клетки были запущены в состояниях перепрограммирования и итеративно обновлялись, чтобы предсказать их будущую экспрессию. Моделируемые клетки дифференцировались в сторону состояний соматических клеток в обоих типах клеток (слева). Расстояние между смоделированными перепрограммированными клетками и состоянием контрольных клеток значительно уменьшилось (справа) после 500 временных шагов в поле скорости РНК (n = 1000 симуляций; p 0,01, t-критерий для одного образца).
Рисунок 2: Идентичность соматических клеток подавляется временным перепрограммированием и повторно приобретается посредством вторичной дифференцировки.(A) Временное перепрограммирование в адипогенных клетках индуцирует непрерывную траекторию перепрограммирования специфичных клеточных состояний. Мы зафиксировали эту траекторию количественно с помощью диффузионной псевдовременной координаты. Скорость РНК (стрелки) предсказывала, что клетки в специфичных для перепрограммирования состояниях дифференцируются в направлении идентичности контрольных клеток. (B) Связанные с плюрипотентностью гены Nanog и Snca были индуцированы в наиболее дистальных состояниях перепрограммирования, в то время как гены идентичности адипоцитов Lpl и Fabp4 были подавлены (значения соответствия GAM, среднее ± 95% ДИ). (C) MSC, полученные из мышц, демонстрируют сходную траекторию состояний перепрограммирования. Скорость РНК аналогичным образом предсказывала, что MSC в специфичных для перепрограммирования состояниях повторно дифференцируются в направлении своей исходной идентичности соматических клеток. (D) Гены, ассоциированные с плюрипотентностью, также индуцировались в дистальных состояниях перепрограммирования MSC, в то время как гены мезенхимальной идентичности Thy1 и Col1a1 подавлены. (E) Программная активность мезенхимальной идентичности, выведенная с использованием классификаторов типов клеток и набора данных атласа клеток мыши, оцененных по псевдовремени (слева) и проецируемых в латентные пространства транскрипции (справа). Программы мезенхимальной идентичности подавлялись в дистальных состояниях перепрограммирования для обоих типов клеток, в то время как энтропия идентичности клеток возрастала (p 0,01, тест Вальда). (F) Обогащенный анализ онтологии генов показывает, что эпителиально-мезенхимальный переход (EMT), внеклеточный матрикс (ECM) и гены передачи сигналов клетки являются основными движущими силами скорости РНК. (G) Траектории состояния клеток моделировались на основе оценок скорости РНК в каждом типе клеток. Смоделированные клетки были запущены в состояниях перепрограммирования и итеративно обновлялись, чтобы предсказать их будущую экспрессию. Моделируемые клетки дифференцировались в сторону состояний соматических клеток в обоих типах клеток (слева). Расстояние между смоделированными перепрограммированными клетками и состоянием контрольных клеток значительно уменьшилось (справа) после 500 временных шагов в поле скорости РНК (n = 1000 симуляций; p 0,01, t-критерий для одного образца).
Мы сначала исследовали эффекты временного перепрограммирования на отдельные маркерные гены для идентичности соматических клеток через псевдовременные траектории. В адипогенных клетках мы обнаружили, что адипогенные гены Lpl и Fabp4 были значительно подавлены в наиболее дистальных перепрограммированных клетках, тогда как ассоциированные с плюрипотентностью гены Nanog, Snca и Fgf13 были активированы (рис. 2B, методы). Мы обнаружили сходный паттерн в MSC, где мезенхимные гены Acta2, Thy1 и Col1a1 подавлялись, в то время как Nanog, Snca и Fgf13 подавлялись (Рис. 2D). Активация Nanog и других генов плюрипотентности после 2-4-дневной индукции SOKM согласуется с предыдущими исследованиями отдельно-клеточного времени перепрограммирования iPSC (Fig. S7) [27]. Важно отметить, что активация Nanog также была предложена как «точка невозврата», в которой некоторые клетки могут не восстановить соматическую идентичность [28]. Затем мы суммировали активность GRN идентичности клеток, используя методы вывода регуляторных сетей [29, 30] и модель классификации идентичности клеток (scNym), обученную на атласе клеток мыши и наших данных [31, 32]. Мы обнаружили, что программы идентичности соматических клеток были значительно подавлены в дистальных состояниях перепрограммирования для обоих типов клеток с использованием обоих подходов к анализу (рис. 2E; рис. S8; p 0,05, тест Вальда для предсказаний идентичности клеток).
Эти результаты контрастируют с предыдущими сообщениями о том, что временное перепрограммирование не подавляет идентичность соматических клеток или не активирует гены плюрипотентности [10, 11]. Чем можно объяснить эту разницу? Предыдущие исследования основывались на измерении небольшого набора маркерных генов в массовых профилях транскрипции. Эти анализы, возможно, не зафиксировали подавление идентичности клеток или активацию генов плюрипотентности в субпопуляции клеток. Таким образом, наше профилирование отдельных клеток может улавливать эффекты экспрессии генов, которые маскируются при массовых измерениях. Подтверждая эту точку зрения, наши измерения отдельных клеток согласуются с исследованиями по отслеживанию клонов, показывающими, что временные импульсы SOKM индуцируют экспрессию Nanog и достаточны для создания плюрипотентных колоний [15]. Наши результаты предполагают, что временное перепрограммирование репрессирует GRN идентичности соматических клеток и активирует GRN поздней стадии плюрипотентности по крайней мере в субпопуляции клеток в нескольких типах клеток, потенциально создавая неопластический риск.
Кратковременно перепрограммированные клетки повторно приобретают соматические идентичности посредством вторичной дифференцировки.
Было высказано предположение, что временная экспрессия факторов перепрограммирования индуцирует временный процесс де-дифференцировки и ре-дифференцировки, так что перепрограммированные клетки временно принимают свойства ранних фаз перепрограммирования, но затем снова обретают свое исходное состояние [33, 28, 10]. На сегодняшний день существует мало доказательств в пользу этой модели в контексте восстановления экспрессии генов в молодости. В дополнение к обширным профилям текущего состояния клетки, отдельно-клеточная RNA-seq предоставляет информацию о будущих состояниях экспрессии генов через скорость РНК, что позволяет нам исследовать направление изменения клеточного состояния по этой траектории [34]. Скорость РНК определяет скорость изменения для каждого гена на основе относительного соотношения сплайсированных и несплайсированных транскриптов при предположении, что несплайсированные транскрипты представляют собой зарождающиеся или вновь транскрибируемые сообщения.
Мы сделали вывод о скорости РНК как для наших экспериментов с адипогенными клетками, так и для MSC и обнаружили, что карты скоростей в обоих типах клеток предполагают, что перепрограммированные клетки повторно дифференцируются к своему базовому состоянию (Рис. 2A, C) [35]. Чтобы идентифицировать гены, которые управляют предсказанными изменениями состояния клеток, мы провели анализ дифференциальной скорости. В обоих типах клеток мы обнаружили, что наборы генов EMT и внеклеточного матрикса были обогащены маркерами скорости у перепрограммированных клеток, подтверждая, что временно перепрограммированные клетки повторно приобретают свои исходные мезенхимальные идентичности (Рис. 2F).
Мы дополнительно количественно оценили карты скоростей РНК, используя фазовое моделирование, метод динамических систем для измерения свойств векторных полей путем моделирования движения точки, движущейся через поле [36, 23, 37]. Здесь мы смоделировали перепрограммированные клетки на каждой карте скоростей и развили их положения на основе предполагаемых скоростей соседних клеток (методы). Мы обнаружили, что фазовые точки, инициализированные в перепрограммированных состояниях, эволюционировали в направлении контроля состояний соматических клеток в обоих типах клеток (рис. 2G). В сочетании с нашим предыдущим результатом, наши анализы отдельных клеток предоставляют первое прямое доказательство того, что восстановление молодой экспрессии генов включает временное подавление GRN идентичности клеток и последующую повторную дифференцировку и реактивацию этих сетей.
Подмножеств факторов Яманака достаточно, чтобы вызвать временные эффекты перепрограммирования.
Исследования временного перепрограммирования в старых клетках в значительной степени исследовали эффекты канонических факторов Yamanaka (SOKM) вместе с дополнительными регуляторами плюрипотентности, которые, как известно, индуцируют плюрипотентность (Nanog, Lin28) [10, 4]. Программы плюрипотентности, активируемые факторами Яманака, содержат положительную обратную связь, поэтому вполне возможно, что подмножество факторов является достаточным для восстановления юношеской экспрессии, как было предложено в недавнем отчете с использованием только SOK [11]. Несколько попыток также продемонстрировали достаточность подмножеств факторов Яманака для генерации iPSC [38, 39, 40, 41], но остается неизвестным, проявляют ли подмножества подобные транскрипционные эффекты только после короткой временной индукции. Как полная SOKM, так и уменьшенная комбинация факторов SOK являются онкогенными, что мотивирует поиск альтернативных стратегий перепрограммирования для восстановления молодой экспрессии генов [18, 42].
Здесь мы исследовали эффекты всех возможных наборов факторов Яманака, используя настраиваемую объединенную систему скрининга (рис. 3A). Наша система вдохновлена другими объединенными системами скрининга и отслеживания [43, 44, 45, 46] и кодирует индуцируемый тетрациклином фактор перепрограммирования вместе с конститутивно выраженным штрих-кодом на отдельном лентивирусе. Мы ввели эти векторы в объединенном формате в двух независимых экспериментах на молодых и старых MSC и выполнили 3-дневный пульс / 3-дневный чейз. Выраженные лентивирусные штрих-коды позволили нам демультиплексировать уникальную комбинацию лентивирусов, инфицирующих каждую клетку in silico (рис. 3A, B; методы). Мы подтвердили точность демультиплексирования путем сравнения с подходом ортогонального демультиплексирования (Рис. S9, Методы).
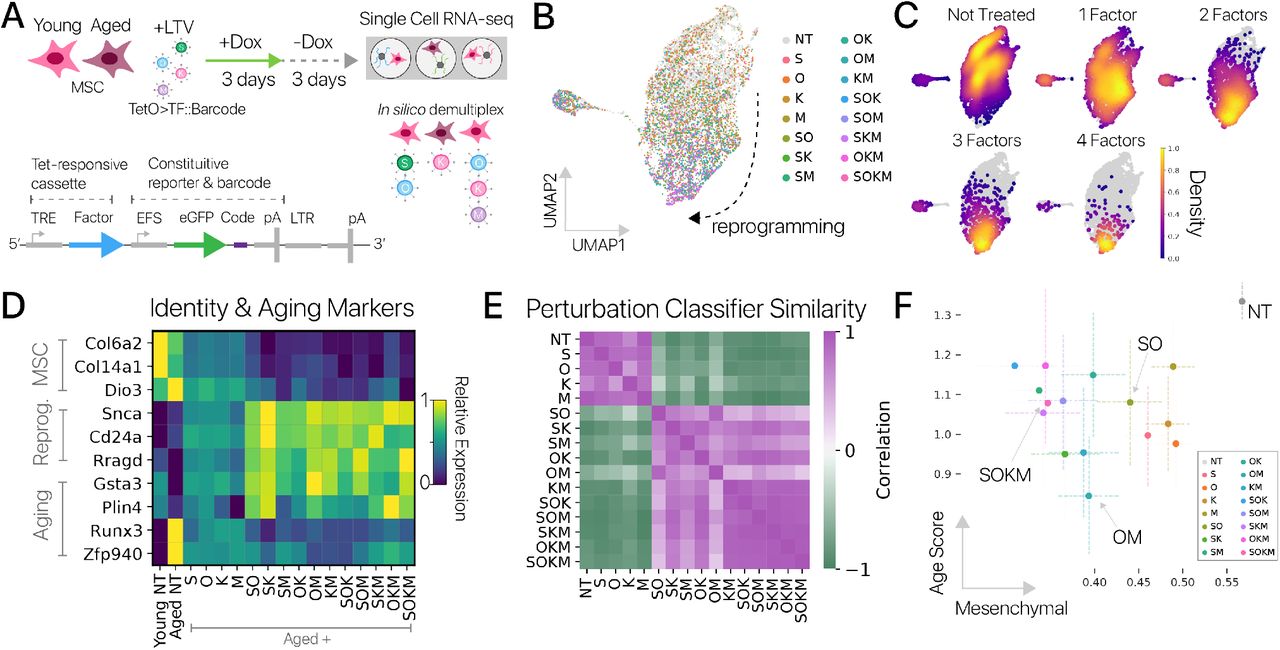 Рисунок 3: Объединенный скрининг показывает достаточность подмножеств факторов Яманака и разделение между омоложением и подавлением идентичности.(A) Схема объединенных скрининговых экспериментов с факторами Яманака. Молодые и старые MSC трансдуцировали лентивирусами, каждый из которых нес один индуцибельный фактор Яманака с выраженными штрих-кодами (внизу). Перепрограммирование индуцировали для 3-дневного пульса / 3-дневного чейза (n = 5 животных на возраст в двух независимых экспериментах). Клетки были профилированы с помощью отдельно-клеточной РНК-seq, и уникальные комбинации факторов Яманака были демультиплексированы in silico на основе выраженных штрих-кодов. (B) Клетки из объединенного скрининга, встроенные с использованием scNym, спроецированные с помощью UMAP и помеченные обнаруженными факторами перепрограммирования (10000+ клеток). (C) Плотность клеток, нарушенных различным количеством факторов перепрограммирования при внедрении UMAP. Комбинации более высокого порядка показывают больший сдвиг транскрипции по сравнению с контрольными клетками. (D) Гены-маркеры MSC (вверху) уменьшаются, а гены-маркеры перепрограммирования (в центре) увеличиваются по мере увеличения комбинаторной сложности (количества уникальных факторов). Гены старения (внизу) также кажутся ближе к юношескому уровню с более сложными искажениями. (E) Матрица сходства между различными комбинациями факторов Яманака, извлеченными из модели классификации возмущений клеток. Сходство вычислялось как корреляция вероятностей предсказания по ячейкам для каждой пары комбинаций. Возмущения низкой сложности и контрольные (NT: необработанные) клетки подобны, в то время как комбинации триплетов очень похожи друг на друга и полный набор факторов Яманака (SOKM). (F) Показатели идентичности мезенхимальных клеток, полученные на основе моделей scNym, и оценки набора генов старения в старых клетках, перепрограммированных с использованием различных комбинаций факторов (средние значения и 95% доверительный интервал). Все комбинации факторов Яманаки, кроме двух, значительно снижали как показатель идентичности мезенхимальных клеток, так и показатель возраста по сравнению со старыми контрольными клетками (NT) (тесты Вальда, p 0,01). Однако возрастные баллы и баллы идентичности не были хорошо коррелированы, предполагая, что подавление идентичности клеток и омоложение не связаны тесно (Спирмен ρ = -0,35, p 0,20).
Рисунок 3: Объединенный скрининг показывает достаточность подмножеств факторов Яманака и разделение между омоложением и подавлением идентичности.(A) Схема объединенных скрининговых экспериментов с факторами Яманака. Молодые и старые MSC трансдуцировали лентивирусами, каждый из которых нес один индуцибельный фактор Яманака с выраженными штрих-кодами (внизу). Перепрограммирование индуцировали для 3-дневного пульса / 3-дневного чейза (n = 5 животных на возраст в двух независимых экспериментах). Клетки были профилированы с помощью отдельно-клеточной РНК-seq, и уникальные комбинации факторов Яманака были демультиплексированы in silico на основе выраженных штрих-кодов. (B) Клетки из объединенного скрининга, встроенные с использованием scNym, спроецированные с помощью UMAP и помеченные обнаруженными факторами перепрограммирования (10000+ клеток). (C) Плотность клеток, нарушенных различным количеством факторов перепрограммирования при внедрении UMAP. Комбинации более высокого порядка показывают больший сдвиг транскрипции по сравнению с контрольными клетками. (D) Гены-маркеры MSC (вверху) уменьшаются, а гены-маркеры перепрограммирования (в центре) увеличиваются по мере увеличения комбинаторной сложности (количества уникальных факторов). Гены старения (внизу) также кажутся ближе к юношескому уровню с более сложными искажениями. (E) Матрица сходства между различными комбинациями факторов Яманака, извлеченными из модели классификации возмущений клеток. Сходство вычислялось как корреляция вероятностей предсказания по ячейкам для каждой пары комбинаций. Возмущения низкой сложности и контрольные (NT: необработанные) клетки подобны, в то время как комбинации триплетов очень похожи друг на друга и полный набор факторов Яманака (SOKM). (F) Показатели идентичности мезенхимальных клеток, полученные на основе моделей scNym, и оценки набора генов старения в старых клетках, перепрограммированных с использованием различных комбинаций факторов (средние значения и 95% доверительный интервал). Все комбинации факторов Яманаки, кроме двух, значительно снижали как показатель идентичности мезенхимальных клеток, так и показатель возраста по сравнению со старыми контрольными клетками (NT) (тесты Вальда, p 0,01). Однако возрастные баллы и баллы идентичности не были хорошо коррелированы, предполагая, что подавление идентичности клеток и омоложение не связаны тесно (Спирмен ρ = -0,35, p 0,20).
Мы извлекли 100 клеток для всех комбинаций факторов Яманака (рис. 3B, C). Чтобы подтвердить эффективность нашей объединенной системы скрининга, мы исследовали влияние каждой комбинации на гены-маркеры перепрограммирования. Мы обнаружили, что гены-маркеры мезенхимы уменьшались в зависимости от комбинаторной сложности (количества уникальных факторов), в то время как гены-маркеры перепрограммирования увеличивались (Рис. 3D). Этот результат согласуется с известной биологией факторов Яманака, где комбинации более высокого порядка более эффективны при перепрограммировании [3]. Затем мы хотели определить, вызывают ли разные комбинации факторов Яманака уникальные эффекты, или каждая комбинация вызывает схожие программы экспрессии генов, различающиеся больше по величине, чем по направлению. Мы исследовали этот вопрос, обучая современную модель классификации идентичности клеток, чтобы различать клетки, возмущенные каждой уникальной комбинацией факторов Яманака (методов) [31]. Комбинации, которые очень похожи, могут быть часто перепутаны моделью, в то время как уникальные комбинации могут быть легко различимы. Чтобы проанализировать наш обученный классификатор, мы извлекли вероятности предсказания для комбинаций для каждой ячейки, а затем вычислили корреляцию этих вероятностей для каждой пары комбинаций в качестве метрики сходства. Мы обнаружили, что возмущения одинаковой комбинаторной сложности очень похожи, в то время как возмущения разной сложности легко различимы. Например, большинство комбинаций триплетов часто путали с полным набором факторов Яманака (рис. 3E). Мы также сравнили дифференциально экспрессируемые гены для каждой комбинации с использованием индекса Жаккара и векторов средней экспрессии с использованием косинусного сходства, получив аналогичные результаты (рис. S10A, B).
Большинство комбинаций факторов Яманака с аналогичной сложностью, таким образом, имеют сходные транскрипционные эффекты при индукции в течение короткого периода времени. Сходство среди комбинаций более высокого порядка может быть связано с активацией общих эффекторов в сети плюрипотентности, даже при отсутствии недостающих факторов, что согласуется с предыдущими сообщениями [38, 39, 40, 41]. Также вероятно, что эндогенная копия отсутствующего фактора активируется остающимся фактором, поскольку факторы Яманака могут активировать друг друга [30] (рис. S10C). Мы не наблюдали значительного повышения регуляции недостающих факторов в наших данных (q 0,1, дифференциальное выражение Монте-Карло), но мы не можем исключить возможность временной активации. Взятые вместе, наши результаты показывают, что для восстановления молодой экспрессии генов не требуется какой-либо один фактор Яманака.
Подмножества факторов Яманака вызывают и омоложение, и подавление идентичности клеток, но в разных пропорциях
Мы задавались вопросом, были ли подавление идентичности клеток и восстановление экспрессии молодых генов сильно связанными при различных комбинациях факторов, так что более сильное восстановление экспрессии молодых генов было бы предсказанием более подавленной программы идентичности. Для исследования мы оценили программу идентификации мезенхимальных клеток с использованием классификатора идентичности клеток и активность набора генов старения, извлеченного из наших экспериментов по MSC клеткам из нашего скрининг- эксперимента [31] (методы). Мы обнаружили, что все комбинации факторов Яманака подавляли программу идентификации клеток и снижали оценку старения (рис. 3). Снижение оценки старения было значительным для всех программ, кроме двух с меньшим количеством ячеек (SOK, OKM; тесты Вальда, p 0,05).
Однако подавление оценки мезенхимальной идентичности и снижение оценки старения не были достоверно коррелированы (Спирмен ρ = -0,35, p 0,20). Этот результат был воспроизведен, когда мы рассматривали каждую из двух независимых экспериментальных партий из нашего скрина отдельно (минимальное q 0,12, максимальное ρ = - 0,1 корреляция Спирмена). Некоторые комбинации снижали оценку старения примерно до той же степени, что и полный набор факторов Яманака, при этом подавляя мезенхимальную идентичность значительно меньше (тест Вальда, p 0,05). Например, комбинация SO, не содержащая онкогенов, имела такое же снижение оценки старения, как и SOKM, но подавляла идентичность мезенхимальных клеток на 54% меньше, чем полный набор. Мы повторили эти эксперименты в меньшем масштабе на адипогенных клетках и получили качественно аналогичные результаты (рис. S11). Этот результат предполагает, что подавление идентичности клеток и восстановление молодой экспрессии генов могут быть разделены. Таким образом, возможно разработать стратегии перепрограммирования, которые восстанавливают юношескую экспрессию, сводя к минимуму риски подавления соматической идентичности.
Временное мультипотентное перепрограммирование улучшает миогенез в старых клетках
Наши результаты с факторами Яманака предполагают, что альтернативные стратегии перепрограммирования могут также восстановить молодую экспрессию. Мы задались вопросом, может ли быть эффективным временное перепрограммирование с мультипотентными факторами перепрограммирования. Чтобы проверить эту гипотезу, мы обратились к системе мультипотентного репрограммирования (в отличие от плюрипотентной системы) в миогенных клетках, по аналогии с регенерацией конечностей у urodele amphibian с использованием фактора транскрипции Msx1. Msx1 / 2, как известно, дедифференцирует миоциты до мультипотентного состояния и имеет задокументированную роль в регенерации пальцев и конечностей [47, 48, 49]. Мы выполнили 3-дневный пульс / 3-4-дневный чейз экспрессии Msx1 с использованием доксициклин-индуцируемой лентивирусной системы в старых миогенных клетках in vitro, а затем профилировали клетки с помощью отдельно-клеточной RNA-seq в двух независимых экспериментах (рис. 4A). Как и в наших экспериментах по плюрипотентному перепрограммированию, мы отсортировали трансген-положительные и отрицательные клетки на основе экспрессии репортера, чтобы захватить контроль и перепрограммировать профили мРНК (рис. 4B, методы).
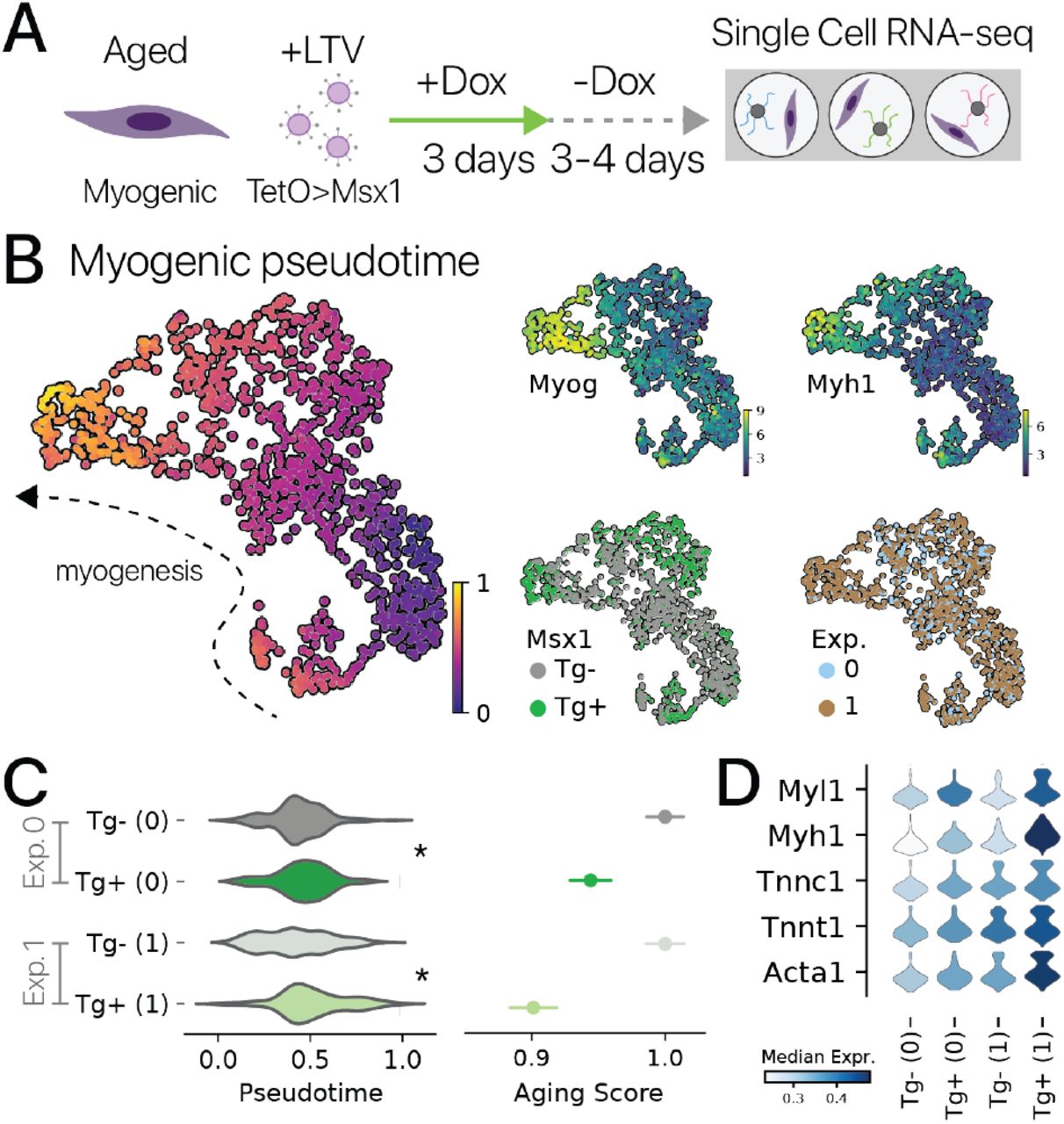 Рисунок 4: Импульсное перепрограммирование Msx1 увеличивает экспрессию миогенных генов в старых миогенных клетках.(A) Схема экспериментов мультипотентного репрограммирования со старыми миогенными клетками, выделенными от (n = 2 и n = 3 животных). (B) Интегрированные профили клеток из двух независимых экспериментов (метки, внизу справа), спроецированные с помощью UMAP. Псевдовременной анализ (слева) обеспечивает количественную оценку состояния миогенной дифференцировки для каждой клетки, подтвержденную анализом маркерного гена (вверху справа). (C) Временное мультипотентное перепрограммирование сдвинуло клетки в значительно более дифференцированное состояние в обоих экспериментах (суммы рангов Вилкоксона, p 0,05). Точно так же временное перепрограммирование значительно снижает сигнатуру транскрипционного старения, извлеченную из предыдущего исследования [23] (среднее ± SEM; t-тест, p 0,01, нормализовано в экспериментах). (D) Миогенные маркерные гены для компонентов саркомера были обогащены временным мультипотентным перепрограммированием в обоих экспериментах (нормализовано внутри экспериментов, q 0,1, Монте-Карло).
Рисунок 4: Импульсное перепрограммирование Msx1 увеличивает экспрессию миогенных генов в старых миогенных клетках.(A) Схема экспериментов мультипотентного репрограммирования со старыми миогенными клетками, выделенными от (n = 2 и n = 3 животных). (B) Интегрированные профили клеток из двух независимых экспериментов (метки, внизу справа), спроецированные с помощью UMAP. Псевдовременной анализ (слева) обеспечивает количественную оценку состояния миогенной дифференцировки для каждой клетки, подтвержденную анализом маркерного гена (вверху справа). (C) Временное мультипотентное перепрограммирование сдвинуло клетки в значительно более дифференцированное состояние в обоих экспериментах (суммы рангов Вилкоксона, p 0,05). Точно так же временное перепрограммирование значительно снижает сигнатуру транскрипционного старения, извлеченную из предыдущего исследования [23] (среднее ± SEM; t-тест, p 0,01, нормализовано в экспериментах). (D) Миогенные маркерные гены для компонентов саркомера были обогащены временным мультипотентным перепрограммированием в обоих экспериментах (нормализовано внутри экспериментов, q 0,1, Монте-Карло).
Мы выделили миогенные клетки как в прогкниторном (Pax7 +), так и в дифференцированном состоянии (Myog +). Это ожидаемо, потому что наша система культивирования in vitro обеспечивает стимул дифференцировки [50] (рис. 4B). Ранее сообщалось, что старые миогенные клетки не дифференцируются в зрелые миоциты так же эффективно, как молодые клетки [51, 23]. Мы обнаружили, что временно перепрограммированные старые клетки были более дифференцированными, чем старые контрольные клетки, на основании псевдотемпоральных распределений, экспрессии маркерных генов и анализа набора генов (рис. 4C, D; рис. S12D). Этот результат был устойчивым к независимому анализу каждого эксперимента (рис. S12A, B, C). Мы также измерили сигнатуру транскрипции старения, полученную в результате предыдущего исследования миогенной дифференцировки, и обнаружили, что временное перепрограммирование значительно снижает оценку старения (рис. 4C), методы, p 0,01) [23]. Сама оценка миогенного старения находится под сильным влиянием программы дифференциации, так что псевдовремя объясняет значительную вариацию в оценке старения (r2 = 0,58, p 0,0001 критерий Вальда). Эти результаты предполагают, что мультипотентное перепрограммирование с помощью Msx1 может частично восстанавливать экспрессию молодых генов в миогенных клетках, подобно факторам Яманака в адипоцитах.
ОБСУЖДЕНИЕ
Старение вызывает широкие изменения экспрессии генов в различных типах клеток млекопитающих, и эти изменения связывались со многими характерными признаками старения [52, 21, 53]. Эксперименты по перепрограммированию клеток показали, что молодые животные могут развиваться из взрослых клеток, а признаки старения могут быть стерты путем полного перепрограммирования до плюрипотентности [2, 3, 5]. Недавние сообщения также предположили, что временной экспрессии SOKM достаточно, чтобы обратить вспять признаки старения и улучшить функцию клеток [9, 10, 11, 12]. Однако было неясно, подавляют ли эти временные вмешательства по перепрограммированию идентичность соматических клеток, активируют ли программы плюрипотентности на поздних стадиях или могут ли альтернативные стратегии перепрограммирования восстановить экспрессию молодых генов.
Здесь мы исследовали эти вопросы, используя измерения экспрессии генов в отдельных клетках, чтобы зафиксировать фенотипическую траекторию временного перепрограммирования и оценить влияние альтернативных методов перепрограммирования. Мы обнаружили, что временное перепрограммирование подавляет идентичность соматических клеток и активирует программы плюрипотентности (рис. 2), вопреки некоторым предыдущим отчетам [10, 11], но согласуется с экспериментами по перепрограммированию iPSC (рис. S7) и исследованиями по отслеживанию клеточных линий при временной экспрессии SOKM. [15]. Делая вывод о скорости РНК [34, 35] и применяя численные инструменты из динамических систем [37], мы также обнаружили, что временно перепрограммированные клетки возвращаются к своим исходным состояниям экспрессии генов после прохождения через промежуточное состояние (Рис. 2). Таким образом, наши профили отдельных клеток выявили временные состояния клеток, которые, вероятно, были замаскированы в предыдущих объемных измерениях, и поддерживают модель, в которой временное перепрограммирование подавляет соматические идентичности, которые позже повторно приобретаются посредством дифференцировки. Для подтверждения этой гипотезы потребуются дальнейшие эксперименты по профилированию популяций отдельных клеток в различные моменты времени во время временного перепрограммирования.
Остается неизвестным, какие из факторов Яманака необходимы для восстановления молодого паттерна экспрессии генов, или какие подмножества могут проявлять отдельные эффекты во время временного перепрограммирования. Предыдущие исследования изучали только один набор факторов за раз, что не позволяло проводить точные сравнения для ответа на эти вопросы [9, 10, 11, 12]. Наши объединенные скрины всех возможных подмножеств факторов Яманака показали, что комбинации из 3-4 факторов Яманака имеют удивительно похожие эффекты, что позволяет предположить, что ни один из факторов не является обязательным для восстановления молодой экспрессии. Комбинации двух факторов Яманака также были более похожи на полный набор SOKM, чем на контроль или однофакторные возмущения, и все комбинации факторов перепрограммирования снижали оценку старческой экспрессии генов (рис. 3). Наши данные демонстрируют, что не требуется единого фактора плюрипотентности, чтобы замаскировать признаки старения, и предполагает, что стратегии перепрограммирования без онкогенов также могут восстанавливать молодую экспрессию генов. Наши эксперименты по мультипотентному перепрограммированию миогенных клеток дополнительно подтверждают это предположение, указывая на то, что молодая экспрессия генов может быть восстановлена даже без активации факторов плюрипотентности (Рис. 4).
Восстановление молодой экспрессии генов может улучшить функцию тканей, подразумевая, что временное перепрограммирование может иметь терапевтический эффект. Однако хорошо известно, что плюрипотентное перепрограммирование является онкогенным процессом [18], даже когда Myc исключен из набора перепрограммирования [42]. Хотя сообщалось, что на основании массовых измерений временное перепрограммирование не подавляет идентичность соматических клеток [9, 10, 11], наши результаты для отдельных клеток показывают, что идентичность соматических клеток подавляется, а GRN на поздней стадии активируются в переходном клеточном состоянии. в нескольких типах клеток (рис. 2). Это повышает вероятность того, что даже временное перепрограммирование может быть онкогенным. Поэтому желательно выявить альтернативные стратегии перепрограммирования для восстановления молодой экспрессии генов с более низким неопластическим риском. С этой целью мы показали, что временное перепрограммирование с различными подмножествами факторов Яманака вызывает транскрипционные эффекты очень сходные с полным набором, и что отдельная мультипотентная система перепрограммирования может обеспечивать молодую экспрессию. Эти результаты указывают на возможность разделить омолаживающие и индуцирующие плюрипотентность эффекты временного перепрограммирования и служат ресурсом для дальнейшего исследования эффектов временного перепрограммирования старых клеток.
МЕТОДЫ
Животные
Мы выделили первичные клетки из подкожной жировой ткани (паховая подушечка) и мышц конечностей молодых (2-4 месяца) и старых (20-30 месяцев) самцов мышей C57Bl / 6. Мыши были заказаны в лабораториях Джексона и выдержаны в Calico Life Sciences, LLC. Молодым животным давали возможность акклиматизироваться в течение не менее 3 недель перед экспериментом. Все эксперименты на животных были одобрены Комитетом по уходу и использованию животных Calico, аккредитованным AAALAC органом. Мы изолировали клетки от двух животных каждого возраста для экспериментов по перепрограммированию SOKM адипоцитов (старые животные, 28 месяцев) и трех животных каждого возраста для экспериментов SOKM мезенхимальных стволовых клеток (старые животные, 23 месяца). каждого возраста для одного независимого объединенного скринингового эксперимента MSC и от двух животных каждого возраста во втором независимом эксперименте (старые животные, возраст 28 месяцев). Мы выделили адипогенные клетки от двух животных каждого возраста в одном объединенном скрининговом эксперименте на адипогенные свойства (старые животные, возраст 28 месяцев) и от трех животных каждого возраста во втором эксперименте (старые животные, возраст 30 месяцев). Для экспериментов с миогенными клетками мы изолировали клетки от двух старых животных (28 месяцев) для первого эксперимента и трех старых животных (30 месяцев) для второго эксперимента.
Изоляция клеток
Мы изолировали пре-адипоцитарные клетки из подкожной жировой ткани, рассекая ткань ножницами на маленькие (1-2 мм) кусочки и выполняя расщепление 0,1% (мас. / Об.) Коллагеназой II (Gibco) в среде DMEM при 37 ° C в течение 1 часа. Суспензии жировых клеток промывают полной средой (10% FBS, DMEM) и инкубируют в 6-луночном планшете в течение ночи , одна лунка на животное. Мы выделили мезенхимальные стволовые клетки из мышечной ткани конечностей, разрезая мышцу на мелкие кусочки ножницами и выполнив две ферментативных варки. Сначала мы варили 0,2% (мас. / Об.) Коллагеназы II в среде DMEM в течение 90 минут, затем промывали клеточные суспензии в буферной воде для варки (F10, 10% лошадиная сыворотка) и проводили вторую инкубацию в 0,4% (мас. / Об.) Диспазе II. (Gibco) на один час. Суспензии обработанных мышечных клеток фильтровали через сетчатый фильтр для клеток 70 мкм, затем через фильтр для клеток 40 мкм и окрашивали антителами, конъюгированными с флуорофором (см. Таблицу S1). Мы отсортировали мезенхимальные стволовые клетки как CD31-, CD45-, PI-, Sca1 + с помощью FACS. МСК помещали в одну лунку каждого 6-луночного планшета для каждого животного и инкубировали в течение 24 часов.
Мы выделили миогенные клетки, выполнив такое же расщепление, которое описано для MSC на мышечной ткани конечностей. После получения суспензий единичных клеток мы выполнили процедуру предварительного посева, инкубируя клетки в лунках, покрытых ECM, полученных от саркомы, в течение 10 минут, затем немедленно перенесли суспензии клеток в новые лунки, покрытые ECM. Этот этап предварительного посева захватывает большинство прикрепленных немиогенных клеток в суспензии и позволяет захватить популяцию миогенных клеток высокой чистоты в последних лунках. Клетки инкубировали в течение ночи в миогенной среде для роста (F10, 20% FBS, 1% Pen / Strep; Gibco, с добавлением 5 нг / мл rFGF2; R&D Systems).
Культура клеток
Адипогенные клетки перед трансдукцией пассировали лентивирусными векторами. Клетки диссоциировали реагентом TrypLE (Gibco) и высевали по 100000 клеток / лунку в 6-луночный планшет, используя отдельные лунки для каждого животного, и трансдуцировали после инкубации в течение ночи. MSC, полученные из мышц, один раз пассировали перед трансдукцией с использованием Try-pLE. МСК высевали из расчета 50 000 клеток / лунку в 6-луночный планшет для экспериментов по полицистронному перепрограммированию и 25 000 клеток / лунку для объединенных скрининговых экспериментов. Для групповых скрининговых экспериментов МСК высевали из расчета 7500 клеток / лунку в 24-луночный планшет, по одному планшету на животное. Для объединенного скрининга адипогенных клеток клетки высевали из расчета 100000 клеток / лунку в 6-луночные планшеты. Миогенные клетки размножали в течение 5-7 дней перед трансдукцией лентивирусов в миогенной ростовой среде и пассировали для посева при 100000 клеток / лунку в 6-луночных планшетах для трансдукции. Миогенные клетки пассировали с буфером для диссоциации клеток (Gibco).
Лентивирусное клонирование и производство
Для экспериментов по полицистронному перепрограммированию мы синтезировали кДНК мышей для Pou5f1, Klf4, Myc и Sox2. Мы удалили стоп-кодоны из всех кДНК, кроме Sox2, и конкатенированных кДНК в полицистрон, используя 2A-пептидные последовательности для конечной открытой рамки считывания O-P2A-K-T2A-M-E2A-S. Мы вставили этот полицистрон OKMS в лентивирусный вектор-переносчик 3-го поколения, фланкированный индуцируемым тетрациклином 5 ’TRE3G-промотором и 3’ ответным элементом wood-chuck промотора. Мы также включили промотор цитомегловируса (CMV), управляемый репортерным транскриптом mCherry 3 ’из WPRE до 5’ конца лентивирусного повтора (LTR). Мы называем этот вектор LTV-Y4TF. Для объединенных скрининговых экспериментов мы синтезировали кДНК для факторов Яманака, за которыми следует eGFP, управляемый коротким промотором EF-1 альфа (EFS) с уникальным 8-мерным штрих-кодом в 3 'UTR, за которым следует внутренний сигнал полиА SV40 в геноме лентивируса. . Мы клонировали каждый трансген фактора Яманака в лентивирусные векторы для переноса с 5 ’промотором TRE3G для управления экспрессией фактора Яманака. Эти разработки были вдохновлены системой отслеживания клонов CellTag [46] и позволяют нам обнаруживать набор факторов Yamanaka, которые трансдуцировали данную клетку на основе восстановления уникальных штрих-кодов, даже несмотря на то, что трансгены фактора Yamanaka экспрессируются только временно. Мы называем эти векторы LTV-S-BC, LTV-O-BC, LTV-K-BC и LTV-M-BC, где BC обозначает 3 ’выраженный штрих-код.
Для экспериментов по мультипотентному перепрограммированию миогенных клеток мы клонировали кДНК Msx1 с разделяемой меткой mCherry Msx1-T2A-mCherry в лентивирусный вектор, управляемый TRE3G, с репортером eGFP, управляемым 3 ’CMV. Мы называем этот вектор LTV-Msx1. Мы использовали три разных лентивирусных вектора-трансактиватора тетрациклина, все несли один и тот же аллель трансактиватора тетрациклина Tet3G, фланкированный разными флуоресцентными репортерами, чтобы обеспечить совместимость с разными векторами перепрограммирования. Для полицистронного перепрограммирования в мышечных МСК мы использовали управляемый ЦМВ вектор Tet3G-T2A-mCerulean (LTV-Tet3G-mCerulean; адаптированный из VectorBuilder cat. VB180123-1018bxq сборкой Гибсона). Для полицистронного перепрограммирования в адипогенных клетках мы использовали вектор, содержащий управляемую EF1a ORF Tet3G, за которой следует репортер eGFP-T2A-PuromycinR, управляемый CMV (LTV-Tet3G-eGFP; VectorBuilder cat. VB900088-2774nkq). Для объединенного скрининга мы использовали вектор, несущий Tet3G, управляемый CMV, за которым следует репортер mCherry, управляемый 3 ’WPRE и CMV (LTV-Tet3G-mCherry; VectorBuilder cat. VB900088-2776tfj).
Мы упаковали лентивирус путем трансфекции клеток HEK293T упаковывающими лентивирусными плазмидами LV-MAX (Gibco) и представляющим интерес вектором переноса, содержащим LTR, с использованием липофектамина 3000 (Gibco). Мы собирали супернатант из вирусных упаковывающих клеток через 24 и 48 часов после трансфекции, очищали супернатант центрифугированием, фильтровали супернатант с помощью фильтров 0,45 мкм и концентрировали очищенный супернатант с использованием реагента для осаждения PEG-it (System-Bio). Лентивирусные конструкции титровали путем трансдукции клеток HEK293T серийными разведениями вируса и измерения частоты экспрессии флуоресцентного репортера через 72 часа. Мы также приготовили лентивирусные частицы через коммерческих поставщиков, которые используют аналогичные протоколы.
Лентивирусная трансдукция
Мы проводили лентивирусные трансдукции с использованием стандартного метода спинфекции для всех типов клеток. Для адипогенных клеток мы смешали соответствующий титр вируса с полной средой для выращивания с добавлением [8 мкг / мл] полибрена и заменили среду для выращивания вирусной суспензией. Мы центрифугировали клетки в планшетах с лунками при 2000 x g в течение 1 часа и инкубировали в течение ночи перед заменой среды для роста. Для МСК мы аналогичным образом добавили вирусную суспензию, центрифугировали клетки при 2000 x g в течение 1 часа и инкубировали клетки в течение ночи перед заменой среды. Для миогенных клеток мы добавили вирусную суспензию и трансдуцировали путем спинфекции в течение одного часа при 1500 x g с полибреновым адъювантом ([8 мкг / мл] в среде).
Переходное полицистронное перепрограммирование
Мы выполнили временное перепрограммирование в адипогенных клетках с использованием LTV-Y4TF и LTV-Tet3G-eGFP. Мы трансдуцировали при MOI 30 в ростовой среде, содержащей полибрен ([8 мкг / мл]), путем центрифугирования в течение 1 часа при 2000 × g. Мы заменили питательную среду после инкубации в течение ночи и начали трехдневный пульс Dox ([4 мкг / мл]), меняя среду каждые 24 часа, после чего следовал трехдневный чейз. Мы выполнили тот же протокол для мышечных МСК с использованием LTV-Y4Tf и LTV-Tet3G-mCerulean каждый при MOI 10. Для мышечных МСК мы также включили контрольную группу, трансдуцированную вирусом, но никогда не подвергавшуюся воздействию Dox. После периода чейза клетки были диссоциированы, и мы отсортировали клетки, экспрессирующие оба репортера LTV (LTV-Y4TF: mCherry, LTV-Tet3G: eGFP или mCerulean), против всех других клеток с помощью FACS. Эта сортировка позволила нам обогатить трансдуцированными клетками и обработанными профилем (положительный двойной трансген) и необработанными (отсутствие хотя бы одного трансгена) в отдельных клетках из одной и той же лунки, что служило контролем in situ.
Скрининг подмножеств факторов Яманака
Мы провели скрининг подмножеств фактора Яманака в двух различных экспериментах на МСК. В первом эксперименте мы высевали клетки в 24-луночные планшеты и доставляли каждую комбинацию факторов Яманака в одну лунку планшета с высоким MOI (MOI = 8) для каждого вируса. Мы доставили вирус-трансактиватор тетрациклина (LTV-CMVTet3G-CMVmCherry) во все лунки (MOI = 8). Перед секвенированием мы пометили сложность каждого возмущения (например, количество уникальных факторов) уникальным олигонуклеотидом, модифицированным холестерином, для сравнения с нашим демультиплексированием in silico. Во втором эксперименте мы засевали клетки в 6-луночные планшеты и доставили все факторы Яманака в пуле в каждую лунку. Мы трансдуцировали по одной лунке на животное как с низким, так и с умеренным значением MOI (MOI = 3, 6) и объединяли клетки перед секвенированием. Два MOI использовались для увеличения представления более сложных возмущений. В обоих экспериментах мы пульсировали факторы Яманака, вводя доксициклин [4 мкг / мл] в течение трех дней, и гоняли в течение 3 дней. Мы аналогичным образом провели объединенный скрининг в двух различных экспериментах на адипогенные клетки. В первом эксперименте мы выделили клетки от двух молодых и двух старых животных и трансдуцировали с помощью MOI 8. Во втором мы выделили клетки от трех молодых и трех старых (30 месяцев) животных и проанализировали с помощью MOI 8. Были выполнены адипогенные скрининга. с 100 000 клеток / лунку в 6-луночном формате.
Временное мультипотентное перепрограммирование в миогенных клетках
Мы провели временное репрограммирование с Msx1 в миогенных клетках в двух независимых экспериментах. В обоих экспериментах мы высевали 100000 миогенных клеток на лунку в 6-луночный планшет в миогенную среду для роста. Мы трансдуцировали клетки с MOI 15 с помощью LTV-Msx1 и LTV-Tet3G-mCherry. Мы инкубировали клетки в течение 12 часов, затем заменили вирусную суспензию свежей средой для выращивания, содержащей [4 мкг / мл] Dox. Мы ежедневно в течение трех дней заменяли носители на Dox, затем начали период чейза, когда использовались носители без Dox. Мы использовали трехдневный чейз для первого эксперимента и четырехдневный чейз для второго. Мы отсортировали клетки, экспрессирующие флуоресцентные репортеры обоих трансгенов против других клеток, с помощью FACS. Эта сортировка позволила нам обогатить трансдуцированными клетками и обработанными профилем (положительный двойной трансген) и необработанными (отсутствие хотя бы одного трансгена) в отдельных клетках из одной и той же лунки, что служило контролем in situ.
Отдельноклеточное секвенирование РНК
Мы выполнили хеширование клеток с олигонуклеотидами, модифицированными холестерином (CMO), чтобы пометить клетки отдельных животных уникальными штрих-кодами. Мы использовали уникальную группу из 1-2 CMO (интегрированных ДНК-технологий) для клеток от каждого животного и помеченных клеток в соответствии с протоколом MULTI-seq [54]. Библиотеки последовательностей РНК одиночных клеток получали с использованием химии экспрессии генов одиночных клеток NextGEM v3.1 (10x Genomics, Плезантон, Калифорния). Клетки эмульгировали с использованием контроллера Chromium (10x Genomics), а затем готовили библиотеки в соответствии с протоколом набора библиотек. Мы запускали молодые и старые клетки на отдельных дорожках прибора 10x для всех экспериментов, чтобы обеспечить высочайшую точность демультиплексирования образцов. Для всех экспериментов, за исключением объединенных скринингов, мы дополнительно запускали трансген-положительные клетки, несущие как вектор перепрограммирования, так и тетрациклин-трансактиватор, и трансген-отрицательные клетки (без хотя бы одного вектора) на отдельных дорожках. Мы добавили аддитивный праймер MULTI-seq на этапе амплификации кДНК, чтобы обеспечить амплификацию штрих-кода CMO, как описано в протоколе MULTI-seq. Для экспериментов по миогенному перепрограммированию мы объединили миогенные клетки с очень разными типами клеток, подготовленными для несвязанных экспериментов, в одной и той же полосе и экстрагировали миогенные клетки in silico для анализа. Мы также объединили миогенные клетки, полученные от разных животных, без штрих-кодирования CMO, чтобы избежать потери клеток для этого редкого типа клеток.
Выравнивание чтения и демультиплексирование клеток
Мы псевдо-выровняли чтения в эталонный геном mm10 с помощью «kallisto» и выполнили демультиплексирование штрих-кода клеток и агрегацию считывания UMI с помощью «kallisto | bus-tools »[55, 56]. Для экспериментов с трансгенными конструкциями мы модифицировали ссылку mm10, чтобы включить лентивирусные геномы в качестве дополнительных хромосом в эталонный геном. Это позволило нам обнаружить присутствие трансгенных транскриптов в наших данных секвенирования. Мы назначили чтение библиотеки MULTI-seq штрих-кодам клеток с помощью «воздушного змея» [57]. Для объединенных скрининговых экспериментов мы дополнительно количественно определили количество штрих-кодов лентивирусного трансгена в каждой клетке с помощью «воздушного змея».
Шумоподавление профиля мРНК, вывод скрытых переменных и интеграция данных
Мы подавили шумы в избыточных профилях мРНК, используя модели scVI [20] с 64 латентными переменными. Мы подбираем модели scVI для 1000 эпох, используя раннюю остановку, чтобы минимизировать нижнюю границу доказательств (ELBO). Для объединенных скрининговых экспериментов MSC мы интегрировали наши объединенные и объединенные эксперименты по трансдукции, вводя пакетные коварианты в модель scVI. Точно так же мы использовали периодические инъекции ковариант для интеграции независимых адипогенных объединенных скрининговых экспериментов и экспериментов по миогенному мультипотентному репрограммированию. Для последнего приложения мы уменьшили размер скрытого пространства до 32 переменных, чтобы избежать переобучения меньшему набору данных. Для миогенных экспериментов мы дополнительно использовали интеграцию «гармонии» при PCA-разложении латентного пространства scVI для учета пакетных эффектов в независимых экспериментах [58]. Чтобы построить общее латентное пространство между адипогенными клетками и МСК, мы приспособили модель scVI к обеим популяциям клеток одновременно. Для всех экспериментов мы построили граф ближайших соседей в латентном пространстве scVI и спроецировали этот граф в двух измерениях для визуализации с помощью UMAP.
Демультиплексирование MULTI-seq
Мы использовали подход «hashsolo» для демультиплексирования счетчиков чтения MULTI-seq как в адипогенных, так и в MSC экспериментах с предварительным распределением [0,02, 0,94, 0,04] для отрицательных, синглетных и дублетных соответственно [59]. Мы вручную проверили классификации «хеш-соло» путем кластеризации профилей ячеек с использованием счетчиков CMO. При необходимости мы скорректировали метки кластера на основе ручной проверки. Мы использовали только вызовы достоверной классификации для анализа конкретных животных.
Объемная последовательность РНК молодых и старых MSC
Мы выполнили объемную последовательность РНК на молодых и старых MSC в трех независимых экспериментах. Клетки собирали у трех молодых (3-4 месяца) и трех старых (20-24 месяцев) животных в каждом эксперименте. В первом и втором эксперименте свежевыделенные клетки культивировали в течение 11-14 дней перед сбором РНК. В третьем эксперименте клетки из второго эксперимента со свежими клетками криоконсервировали в замораживающей среде для восстановления культур клеток (Gibco), затем размораживали и культивировали в течение 7 дней до сбора РНК. РНК выделяли из всех клеток, используя набор Zymo Quick RNA (Zymo Research), и библиотеки РНК-seq получали с помощью набора NEB-Next Ultra II Directional RNA Library Prep Kit (New England Biolabs). Библиотеки секвенировали на Illumina NovaSeq. Мы псевдо-выровнены чтения с помощью kallisto [55] и количественно оценили дифференциальное выражение с помощью sleuth [60]. Мы извлекли сигнатуру гена старения, выбрав гены, которые изменялись с возрастом ниже свободного порога FDR (q 0,4) как в экспериментах с свежевыделенными клетками, так и в экспериментах с криогенно консервированными клетками. Эта процедура дала сигнатуру 198 генов старения, которые мы использовали для оценки экспрессии генов старения в нашем эксперименте по перепрограммированию MSC SOKM.
Оценка вклада ковариат в общую вариабельность
Мы оценили вклад экспериментальных ковариат (возраст, обработка трансгена) в общую вариацию, наблюдаемую в наших экспериментах, с помощью дисперсионного анализа. Мы рассматривали латентное кодирование scVI как набор переменных отклика и подбирали линейные модели для каждой переменной вида zj ∼age + treatment + age: treatment, где zj - латентная переменная. Мы также оценили вклад животного происхождения для каждой клетки с использованием специфичных для животных меток, полученных в результате демультиплексирования MULTI-seq. Мы выполнили ANOVA, как указано выше, с метками, специфичными для животных, с использованием моделей формы zj ∼ возраст * лечение + животное, где «животное» - это метка, специфичная для каждой комбинации «возраст: обработка», потому что мы использовали комбинации «возраст: обработка» отдельно реакции на подготовку библиотеки. Мы включили «отрицательные» классификации из «hashsolo» в эту регрессию на случай, если сбой хеширования CMO зафиксировал структурированные вариации.
Дифференциальная экспрессия
Мы выполнили тестирование дифференциальной экспрессии по бинарным контрастам, оценив логарифмические распределения изменений для каждого гена с использованием приближений Монте-Карло из апостериорного распределения scVI [61]. Мы оценили коэффициент ложного обнаружения (FDR) дифференциальной экспрессии как долю выборок Монте-Карло, которые не показали логарифмических изменений выше минимального порога (| log2 a / b | ≥0,5). Чтобы проверить непрерывные ковариаты и эффекты взаимодействия типа клетки: перепрограммирование, мы использовали логистические / гауссовские модели препятствий, вдохновленные MAST [62] для отдельных генов, и логистические модели для оценок генной программы на единичном интервале [0, 1], как описано ранее [23] ]. Мы выполнили FDR-контроль для моделей MAST с помощью процедуры Бенджамини-Хохберга [63]. Мы выполнили тестирование дифференциальной экспрессии на псевдовременных ковариатах, используя модели вида «Gene ∼Pseudotime». Мы протестировали тип клеток: взаимодействие перепрограммирования с моделями формы «Ген ∼ Возраст + Тип клетки + Обработка + Тип клетки: Обработка».
Анализ обогащения набора генов
Мы выполнили анализ обогащения для терминов генной онтологии с помощью Enrichr [64]. Мы выполнили ранговый анализ обогащения набора генов (GSEA), используя реализацию «быстрого GSEA» алгоритма GSEA [24] и наборы генов, извлеченные из MSigDB [65].
Обобщенное аддитивное моделирование экспрессии генов в псевдовремени
Мы подбираем обобщенные аддитивные модели (GAM), чтобы выразить отношения между генами или генными программами и непрерывные псевдовременные координаты. Мы подбираем гауссовские GAM в форме «Gene ∼ Pseudotime», используя шесть кубических сплайнов через структуру PyGAM [66]. Мы представляем тренды выражений за псевдовремя, используя среднее значение прогнозов GAM вместе с 95% доверительным интервалом.
Оценка степени старения в транскрипционном пространстве
Мы оценили величину старения как разницу между молодыми и старыми популяциями клеток. Мы оценили эту разницу между двумя популяциями, используя максимальное среднее расхождение (MMD), вычисленное на латентных переменных scVI. Скрытые переменные scVI фиксируют выразительное низкоразмерное представление экспрессии генов и поэтому полезны для этой задачи. Мы вычислили MMD в нашем ранее описанном пакете «scmmd» по серии случайных выборок начальной загрузки, чтобы гарантировать надежность [23]. Вкратце, мы отобрали n = 300 клеток из каждой популяции и вычислили MMD на каждой из 500 итераций. Мы выполнили эти сравнения MMD, чтобы сравнить молодые, необработанные клетки как со старыми, необработанными клетками, так и с старыми перепрограммированными клетками. Мы оценили значимость изменений в MMD с помощью теста Wilcoxon Rank Sum для итераций начальной загрузки.
Скоростной анализ РНК
Мы подсчитали оценочные доли сплайсированных и несращенных считываний для каждого транскрипта на основе сопоставления считываний с экзонами и интронами с использованием «kallisto | bustools »к эталонному геному mm10 из Ensembl [55, 56]. Мы оценили скорость РНК [34], используя стохастическую модель, реализованную в «scvelo» [35].
Псевдотемпоральный вывод траектории
Мы вывели псевдовременные траектории для адипогенных клеток и МСК с помощью «scvelo». Вкратце, мы построили граф ближайших соседей в пространстве вложения и взвешенные ребра в графе на основе направленности векторов скорости РНК, так что соседи на пути вектора скорости РНК получили более высокие вероятности перехода. Затем мы соединили эту матрицу с методом диффузионного псевдовременного вывода [67]. Мы вывели псевдовременные траектории миогенных клеток с помощью диффузионного псевдовременного анализа на графе ближайших соседей, построенном в пространстве компонентов диффузии. Мы выбрали корневые клетки из верхнего дециля экспрессии Snai2 (примитивного маркерного гена).
Фазовое моделирование в полях скоростей РНК
Мы выполнили фазовое моделирование в полях скоростей РНК, используя наш ранее описанный пакет «velodyn» [23]. И для адипогенных клеток, и для МСК мы инициализировали n = 1000 фазовых точек внутри временно перепрограммированной популяции клеток в положениях x0 и развили их положения на основе векторов скорости РНК их соседей для t = 500 временных шагов с размером шага η = 0,5. Мы использовали правило обновления
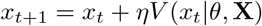
где мы обновляем текущую позицию xt до новой позиции xt + 1, используя функцию обновления V (xt | θ, X), которая выводит вектор скорости vt из k-ближайших соседей с заданными параметрами (k, η). Мы параметризуем V как случайную функцию выборки из многомерного взвешенного гауссовского распределения ? (µ ∗, Σ ∗), где соседи взвешиваются на основе расстояния в пространстве вложения от точки запроса.
Возрастная классификация клеток
Мы обучили модели вариационных автоэнкодеров с полууправлением, используя структуру scANVI, чтобы различать возраст клеток [68]. Перед обучением модели мы разделяем наши данные на обучающий набор (80%) и тестовый набор (20%) со стратификацией. Мы обучили модели scANVI для 100 неконтролируемых эпох (только потеря реконструкции) и 300 частично контролируемых эпох (потеря реконструкции и классификации), используя 10% обучающего набора в качестве данных проверки для ранней остановки. Мы оценили производительность модели на основе точности классификаций в проведенном наборе тестов, невидимой во время обучения модели.
Встраивание возмущений факторами перепрограммирования с помощью scNym
Мы подбираем модели классификации идентичности клеток scNym, чтобы различать МСК, временно перепрограммированные с помощью различных подмножеств фактора Яманака [31]. Мы выделили сбалансированный обучающий набор из полных объединенных данных скрининга MSC путем извлечения n = 100 клеток на комбинацию факторов. Мы относились ко всем оставшимся ячейкам как к испытательному набору. Мы обучили модель scNym на обучающем наборе, используя nhidden = 128 скрытых единиц на слой, период терпения 30 эпох до ранней остановки, максимальное количество эпох 150 и настройки по умолчанию для всех остальных параметров.
Впоследствии мы предсказали комбинации факторов для всех клеток в объединенных скрининговых экспериментах MSC. Мы спроектировали активации встраивания scNym для визуализации с использованием UMAP. Мы вычислили корреляцию предсказаний scNym для каждой комбинации факторов, вычислив корреляции между столбцами матрицы вероятностей предсказанного класса scNym ŷCells × Combinations.
Оценка программ идентификации клеток с помощью scNym
Мы использовали модели scNym для оценки активности конкретных программ клеточной идентификации в временно перепрограммированных клетках. Мы подбираем модели противостояния с полууправлением, используя Tabula Muris в качестве обучающего набора и наши временно перепрограммированные ячейки в качестве целевого набора данных. Мы использовали аннотации «класса онтологии ячеек» в Tabula Muris в качестве меток классов. Мы подбираем модели до 200 эпох и использовали раннюю остановку на проверочном наборе, взятом из обучающих данных, чтобы выбрать наиболее эффективную модель. Мы подбираем отдельные модели для эксперимента по перепрограммированию адипогенных SOKM, эксперимента по перепрограммированию MSC SOKM и объединенного экрана MSC.
Мы использовали обученные модели как для получения встраивания, так и для прогнозирования идентичности клеток из обучающего набора Tabula Muris в наших временно перепрограммированных ячейках. Мы суммировали вероятность нескольких сходных идентичностей клеток, чтобы создать оценку «мезенхимальной идентичности» для каждой клетки (идентичности: «мезенхимальная стволовая клетка», «мезенхимальная клетка», «стромальная клетка»). Мы вычислили энтропию идентичности ячейки как энтропию Шеннона вектора вероятности идентичности ячейки, предсказанного scNym.
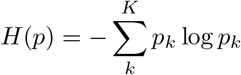
где k ∈ K - индексы классов, p - вектор единичной вероятности, а H (·) - функция энтропии.
Оценка экспрессии генов старения в объединенных скрининговых экспериментах
Чтобы сравнить влияние различных комбинаций факторов Яманака на особенности старения, мы разработали «Шкалу старения», основанную на генах, дифференциально экспрессируемых с возрастом в МСК. Сначала мы идентифицировали гены, которые значительно дифференциально экспрессировались (q 0,10) в одном и том же направлении в контрольных МСК как для нашего эксперимента по полицистронному перепрограммированию, так и для объединенных скринингов. Мы сделали пересечение этого набора с набором генов, которые показали значительное восстановление экспрессии генов молодости в любой из комбинаций фактора Яманака. Это дало набор из 228 генов старения, на которые влияет репрограммирование. Мы получили отдельные оценки AgeUp и AgeDown, используя стандартный метод оценки набора генов для генов с повышенной и пониженной регуляцией соответственно [69]. Затем мы взяли разницу между этими показателями как показатель старения: показатель старения = AgeUp -AgeDown. Мы использовали аналогичную процедуру для объединенных скрининговых экспериментов на адипогенные факторы, чтобы получить набор из 801 гена старения и соответствующий показатель старения. Чтобы количественно оценить влияние каждой комбинации на возрастную оценку, мы использовали линейную регрессию в форме: Оценка старения ∼ Комбинация + Возраст с использованием старых клеток для всех обработок и только контрольных (NT) молодых клеток в качестве данных наблюдений. Мы исключаем молодые обработанные клетки из регрессии, чтобы коэффициенты для каждой комбинации отражали влияние комбинации на старые клетки. Мы извлекли коэффициенты и их доверительные интервалы для сравнения между комбинациями (рис. 3F).
Оценка старческой экспрессии генов в экспериментах по миогенному мультипотентному перепрограммированию
Мы повторно проанализировали общедоступные профили последовательностей РНК одиночных клеток молодых (3 месяца) и старых (20 месяцев) миогенных клеток после дифференцировки in vitro для определения сигнатуры гена старения (GEO: GSE145256) [23]. Мы извлекли 329 генов, которые были изменены более чем в 2 раза между молодыми и старыми миогенными клетками с коэффициентом ложного обнаружения q 0,10 (тест Wilcoxon Rank Sum, контроль Benjamini-Hochberg FDR). Затем мы сгенерировали оценки AgeUp и AgeDown, как описано выше, чтобы построить оценку старения. Для наглядности мы перенесли оценку старения в процентильный интервал [3, 97].
Повторный анализ времени перепрограммирования ИПСК
Мы повторно проанализировали данные предыдущего исследования последовательности РНК одной клетки эмбрионального фибробласта мыши с целью перепрограммирования ИПСК (GEO: GSE122662) [27]. Мы обучили модель scVI шумоподавлению данных, изучению скрытых переменных и выполнению дифференциального выражения, как указано выше.
Повторный анализ старения жировой ткани Tabula Muris Senis
Мы повторно проанализировали данные исследования одноклеточной РНК Tabula Muris Senis старения мышей в жировой ткани (GEO: GSE149590) [21]. Мы нормализовали данные экспрессии с помощью логарифмической трансформации (CountsPerMillion + 1), выбрали сильно вариабельные гены и встроили клетки с помощью UMAP на графе ближайших соседей, полученном с помощью PCA. Мы выполнили дифференциальное выражение с двухвыборочным t-критерием, используя scanpy [70].
Вклад авторов
AR разработала и провела эксперименты по молекулярной биологии и внесла свой интеллектуальный вклад. CZ и JZS проводили эксперименты на животных. JP провел цитометрические эксперименты. TV провел массовые эксперименты с последовательностью РНК. Исследование под руководством Г.К. CK внес интеллектуальный вклад и отредактировал статью. JCK задумал исследование, спроектировал эксперименты, провел эксперименты с культурами клеток, молекулярной биологией, перепрограммированием и секвенированием РНК отдельных клеток, проанализировал данные, руководил исследованиями и написал статью.
Дополнительные рисунки
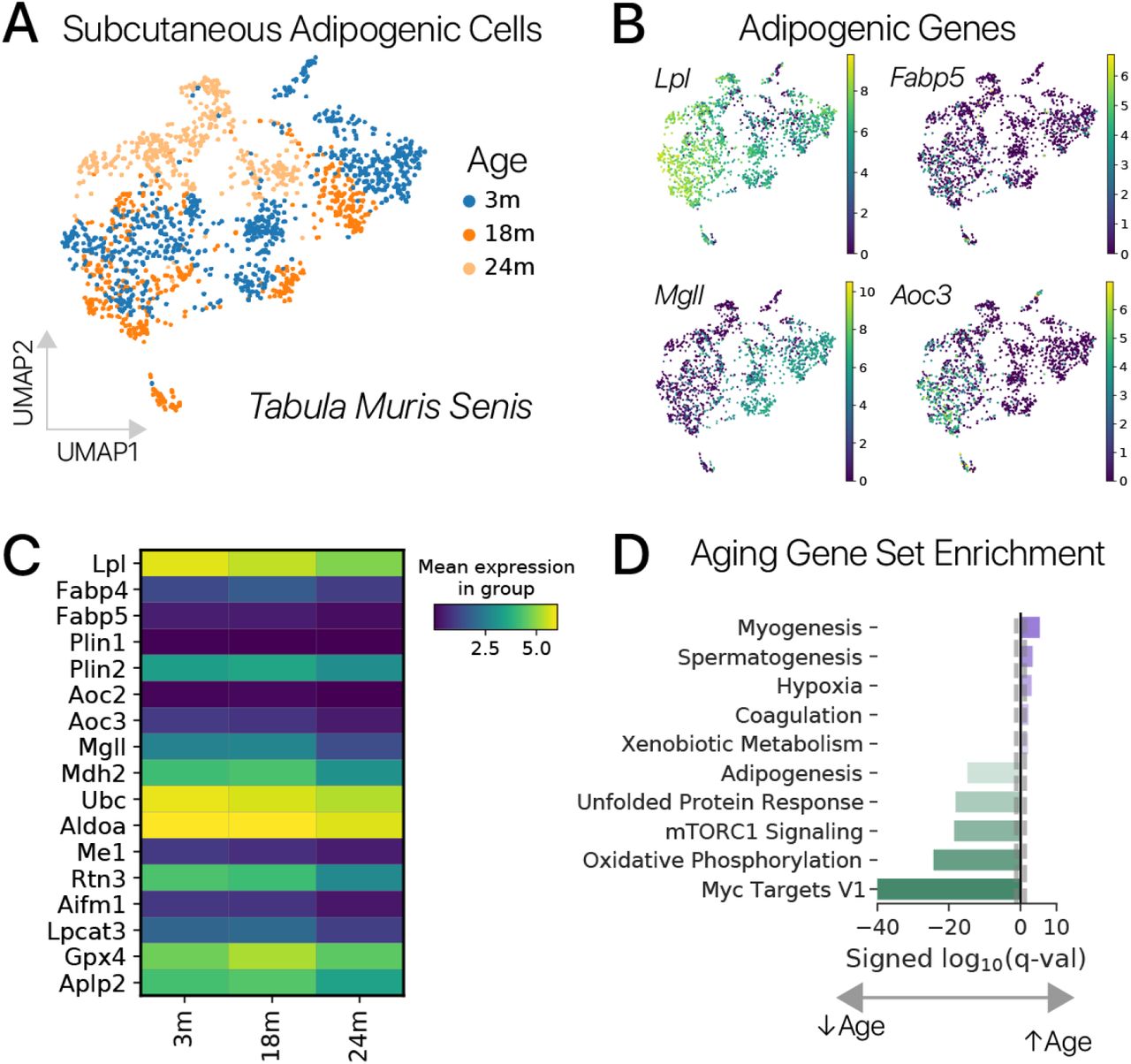 Рисунок S1: адипогенные клетки in vivo демонстрируют сходные черты старения с адипогенными клетками in vitro.(A) Адипогенные клетки из подкожной жировой ткани в Tabula Muris Senis [21], залитые UMAP, окрашены по возрасту. Молодые клетки (3 месяца, месяцы) и старые клетки (24 месяца) четко разделяются в пространстве транскрипции, аналогично нашим данным in vitro. (B) Экспрессия нескольких адипогенных генов ограничена молодыми клетками. (C) Многие адипогенные гены демонстрируют значительно сниженную экспрессию с возрастом, аналогично результатам in vitro (q 0,05, t-тест). (D) Анализ обогащения набора генов для генов, значительно изменившихся в возрасте от 3 месяцев (3 месяца) до 24 месяцев (24 месяца), показывает сильное подавление программ адипогенеза, окислительного фосфорилирования и развернутого белкового ответа (Hallmark MSigDB, q 0,01, точное определение Фишера).
Рисунок S1: адипогенные клетки in vivo демонстрируют сходные черты старения с адипогенными клетками in vitro.(A) Адипогенные клетки из подкожной жировой ткани в Tabula Muris Senis [21], залитые UMAP, окрашены по возрасту. Молодые клетки (3 месяца, месяцы) и старые клетки (24 месяца) четко разделяются в пространстве транскрипции, аналогично нашим данным in vitro. (B) Экспрессия нескольких адипогенных генов ограничена молодыми клетками. (C) Многие адипогенные гены демонстрируют значительно сниженную экспрессию с возрастом, аналогично результатам in vitro (q 0,05, t-тест). (D) Анализ обогащения набора генов для генов, значительно изменившихся в возрасте от 3 месяцев (3 месяца) до 24 месяцев (24 месяца), показывает сильное подавление программ адипогенеза, окислительного фосфорилирования и развернутого белкового ответа (Hallmark MSigDB, q 0,01, точное определение Фишера).
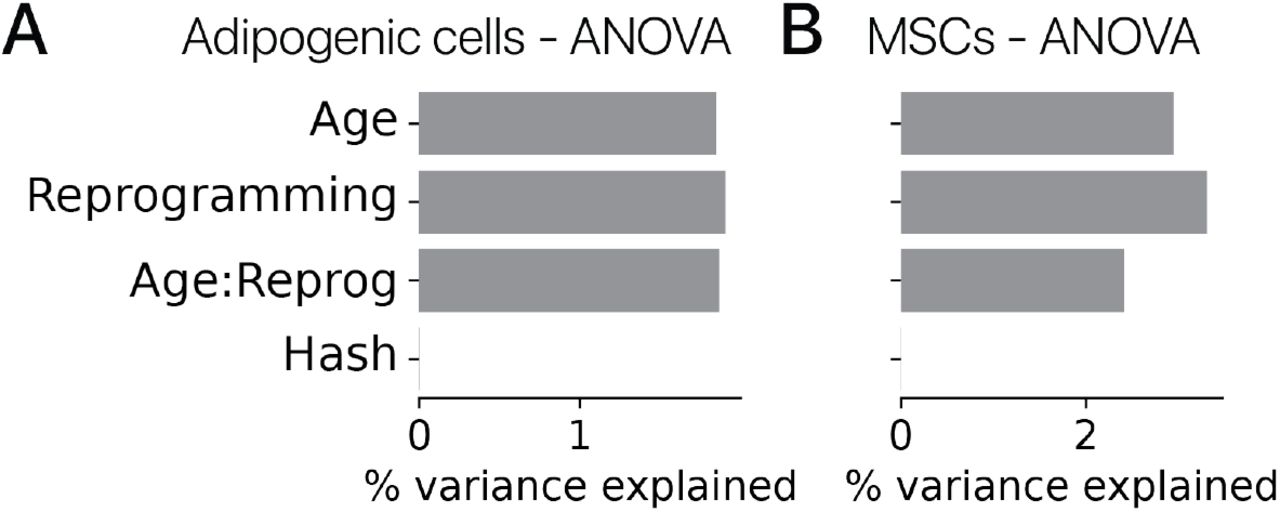 Рисунок S2: Возраст и эффекты перепрограммирования являются доминирующими источниками изменений в временно перепрограммированных клетках.(A) ANOVA приводит к временно перепрограммированным адипогенным клеткам, где оставшаяся вариация не объясняется показанными ковариатами. (B) ANOVA приводит к временно перепрограммированным МСК. В обоих типах клеток возраст и лечение перепрограммированием являются основными источниками объясненных вариаций, в то время как животное (хэш) является второстепенным фактором.
Рисунок S2: Возраст и эффекты перепрограммирования являются доминирующими источниками изменений в временно перепрограммированных клетках.(A) ANOVA приводит к временно перепрограммированным адипогенным клеткам, где оставшаяся вариация не объясняется показанными ковариатами. (B) ANOVA приводит к временно перепрограммированным МСК. В обоих типах клеток возраст и лечение перепрограммированием являются основными источниками объясненных вариаций, в то время как животное (хэш) является второстепенным фактором.
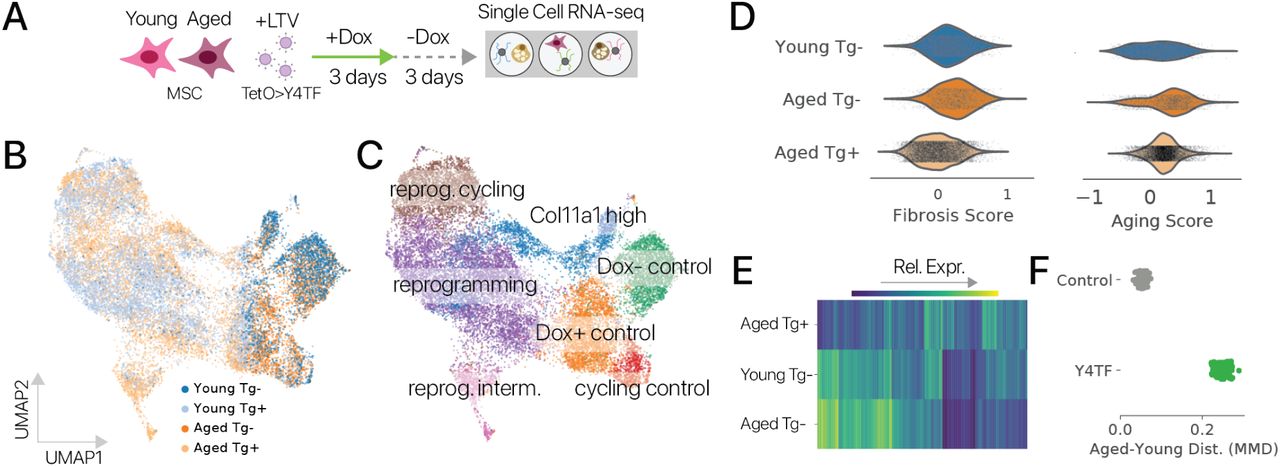 Рисунок S3: Временное перепрограммирование в мышечных МСК ремоделирует экспрессию генов.(А) Схема эксперимента. MSC, полученные из мышц, трансдуцировали с помощью индуцируемой тетрациклином полицистронной кассеты фактора Яманака и временно перепрограммировали с помощью 3-дневного импульса Dox и 3-дневного Dox-чейза. (B) Репрограммированные клетки (Tg +) четко отделяются от контрольных клеток (Tg-) в латентном пространстве транскрипции, в то время как возраст является меньшим источником вариаций. (C) Профили отдельных клеток разрешают различные состояния клеток, вызванные перепрограммированием. (D) Временное перепрограммирование улучшает вызванное старением увеличение наборов генов фиброза и оценку генов старения, полученную в экспериментах с объемной последовательностью РНК. (E) Молодежная экспрессия генов восстанавливается в тысячах генов посредством временного перепрограммирования, несмотря на большую величину эффектов перепрограммирования, ортогональных оси старения. (F) Временное перепрограммирование увеличивает максимальное среднее расхождение между молодыми контрольными клетками и старыми клетками, предполагая, что многие эффекты перепрограммирования ортогональны оси старения.
Рисунок S3: Временное перепрограммирование в мышечных МСК ремоделирует экспрессию генов.(А) Схема эксперимента. MSC, полученные из мышц, трансдуцировали с помощью индуцируемой тетрациклином полицистронной кассеты фактора Яманака и временно перепрограммировали с помощью 3-дневного импульса Dox и 3-дневного Dox-чейза. (B) Репрограммированные клетки (Tg +) четко отделяются от контрольных клеток (Tg-) в латентном пространстве транскрипции, в то время как возраст является меньшим источником вариаций. (C) Профили отдельных клеток разрешают различные состояния клеток, вызванные перепрограммированием. (D) Временное перепрограммирование улучшает вызванное старением увеличение наборов генов фиброза и оценку генов старения, полученную в экспериментах с объемной последовательностью РНК. (E) Молодежная экспрессия генов восстанавливается в тысячах генов посредством временного перепрограммирования, несмотря на большую величину эффектов перепрограммирования, ортогональных оси старения. (F) Временное перепрограммирование увеличивает максимальное среднее расхождение между молодыми контрольными клетками и старыми клетками, предполагая, что многие эффекты перепрограммирования ортогональны оси старения.
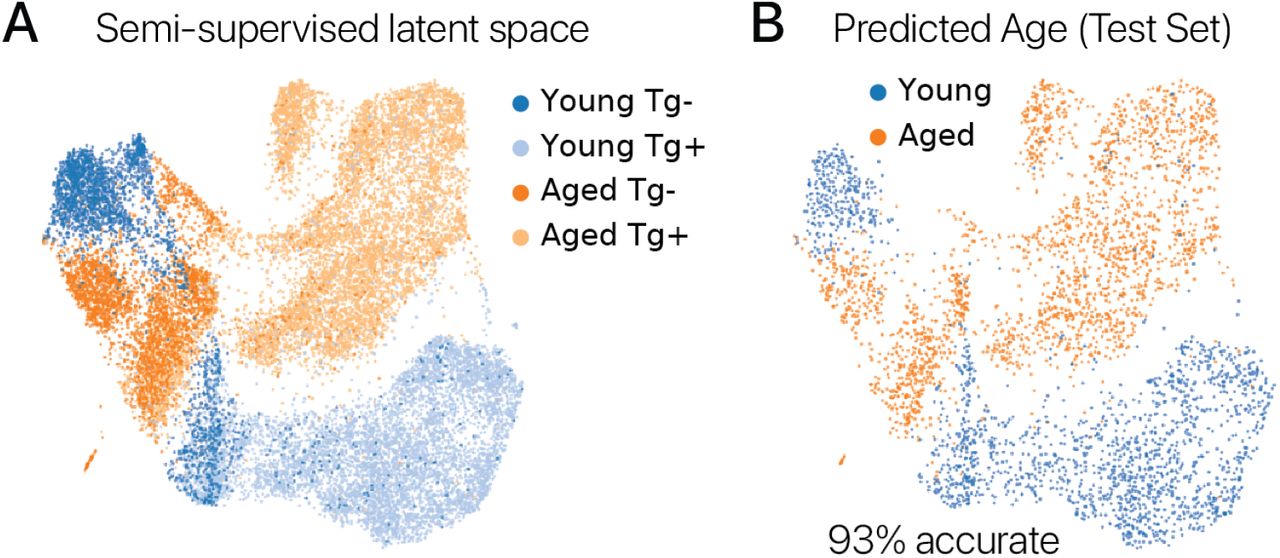 Рисунок S4: Полу-контролируемые классификаторы старения демонстрируют сегрегацию МСК по возрасту в транскрипционном пространстве.(A) MSC, встроенные в скрытое пространство с использованием полууправляемой вариационной модели автокодера. Мы обучили вариационный автоэнкодер совместно реконструировать транскриптомы и классифицировать возраст клеток, изучая представление, которое разделяет молодые и старые клетки в латентном пространстве. (B) Ячейки из тестового набора, спроецированные в частично контролируемое скрытое пространство, окрашенное предсказаниями класса. Мы обнаружили, что предсказания возраста были точными на 93%, подтверждая, что особенности старения фиксируются на уровне транскрипции.
Рисунок S4: Полу-контролируемые классификаторы старения демонстрируют сегрегацию МСК по возрасту в транскрипционном пространстве.(A) MSC, встроенные в скрытое пространство с использованием полууправляемой вариационной модели автокодера. Мы обучили вариационный автоэнкодер совместно реконструировать транскриптомы и классифицировать возраст клеток, изучая представление, которое разделяет молодые и старые клетки в латентном пространстве. (B) Ячейки из тестового набора, спроецированные в частично контролируемое скрытое пространство, окрашенное предсказаниями класса. Мы обнаружили, что предсказания возраста были точными на 93%, подтверждая, что особенности старения фиксируются на уровне транскрипции.
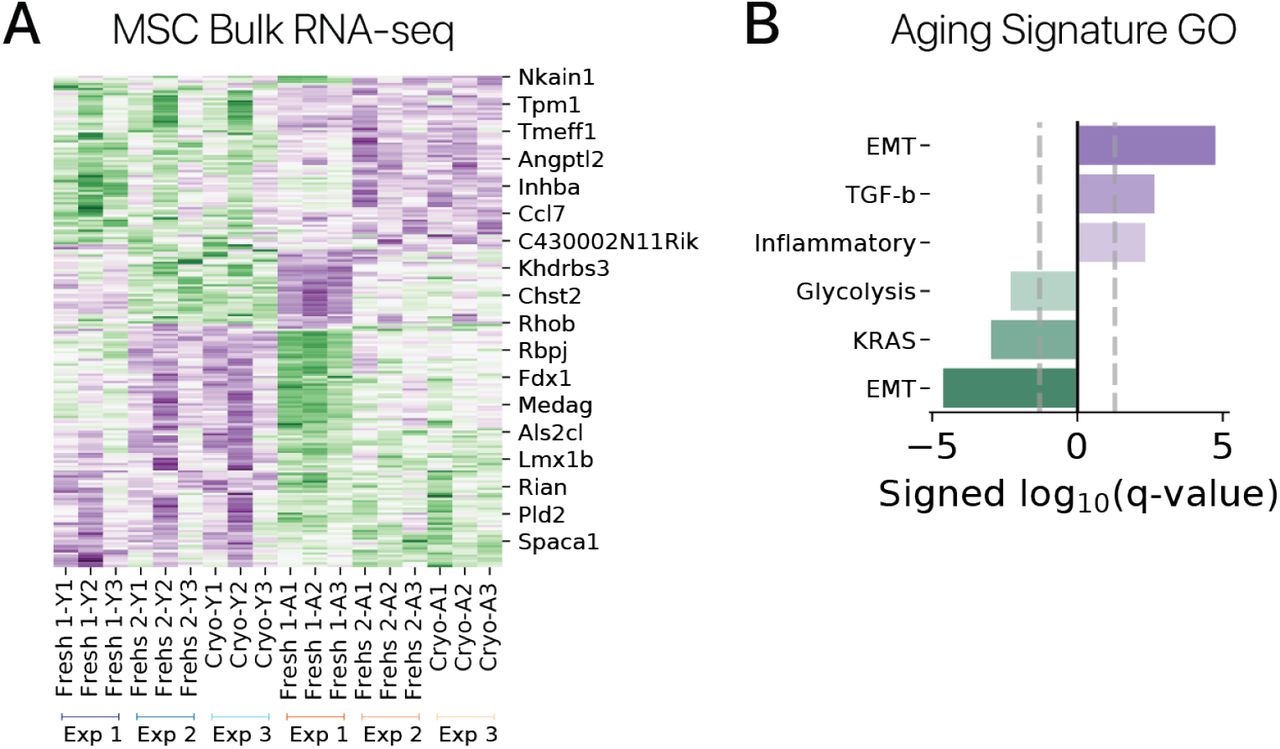 Рисунок S5: Bulk RNA-seq показывает воспроизводимую транскрипционную сигнатуру старения в МСК.(A) Профили массовых последовательностей РНК молодых (Y) и старых (A) МСК, собранные в трех отдельных экспериментах. Показаны гены со значительными эффектами старения во всех трех экспериментах. Мы использовали эти наборы генов для получения «оценки старения» для нашего эксперимента по временному перепрограммированию (рис. S3D). «Свежие» образцы были приготовлены из свежевыделенных МСК после периода культивирования in vitro, в то время как образцы «Крио» были приготовлены после криоконсервации и последующего культивирования. (B) Анализ обогащения онтологии генов для наборов генов MSigDB Hallmark. Гены эпителиально-мезенхимального перехода (EMT) сильно обогащены для возрастных изменений. Как воспалительные гены, так и сигнальный фактор фиброза TGF-β возрастают с возрастом.
Рисунок S5: Bulk RNA-seq показывает воспроизводимую транскрипционную сигнатуру старения в МСК.(A) Профили массовых последовательностей РНК молодых (Y) и старых (A) МСК, собранные в трех отдельных экспериментах. Показаны гены со значительными эффектами старения во всех трех экспериментах. Мы использовали эти наборы генов для получения «оценки старения» для нашего эксперимента по временному перепрограммированию (рис. S3D). «Свежие» образцы были приготовлены из свежевыделенных МСК после периода культивирования in vitro, в то время как образцы «Крио» были приготовлены после криоконсервации и последующего культивирования. (B) Анализ обогащения онтологии генов для наборов генов MSigDB Hallmark. Гены эпителиально-мезенхимального перехода (EMT) сильно обогащены для возрастных изменений. Как воспалительные гены, так и сигнальный фактор фиброза TGF-β возрастают с возрастом.
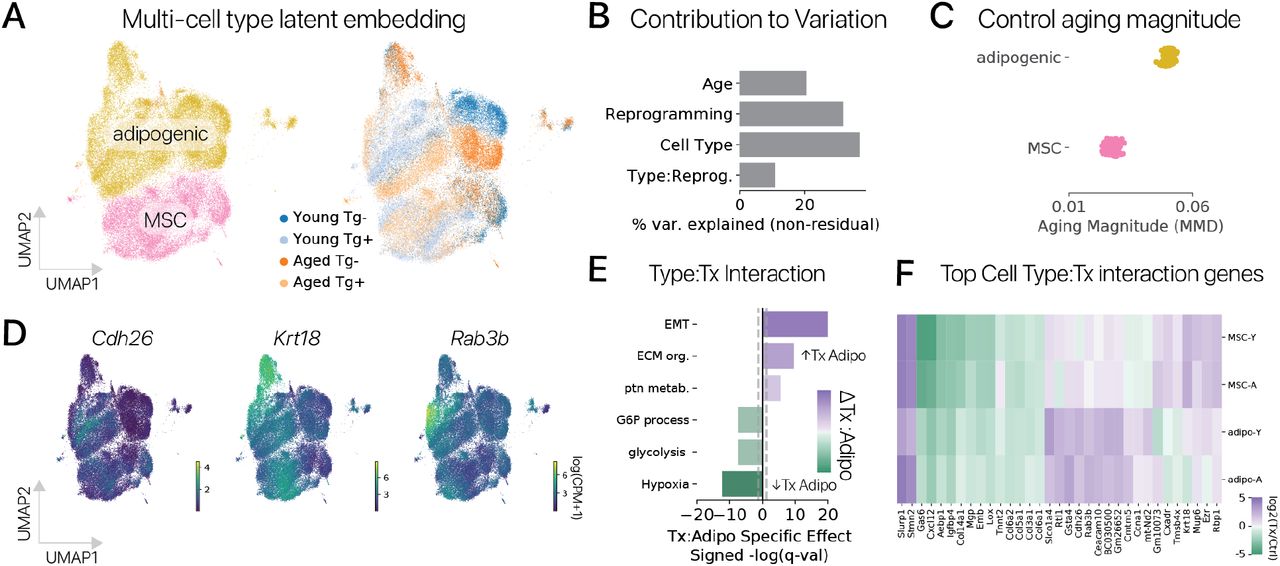 Рисунок S6: Идентичность клетки определяет эффекты временного перепрограммирования.(A) Кратковременно перепрограммированные адипогенные клетки и МСК из наших полицистронных экспериментов были встроены в совместное латентное пространство с scVI, чтобы обеспечить прямое сравнение размеров эффекта. (B) ANOVA показал, что взаимодействия типа клетки: перепрограммирование составляют примерно 11% от общей неостаточной вариации, объясняемой ковариатами. (C) Мы повторно вычислили величины старения по максимальному среднему расхождению для контрольных адипогенных клеток и МСК в суставном латентном пространстве, чтобы учесть прямое сравнение. Мы обнаружили, что степень старения была значительно выше в адипогенных клетках (p 0,01, критерий суммы рангов Вилкоксона). (D) Мы выполнили дифференциальную экспрессию, чтобы идентифицировать гены со значительными взаимодействиями типа клетки: перепрограммирование. Качественный анализ экспрессии генов в латентном пространстве выявляет изменения эффектов, специфичных для типов клеток, по траекториям репрограммирования. (E) Мы использовали анализ генной онтологии, чтобы исследовать наиболее значимые взаимодействия типа клетки: перепрограммирование. Наборы генов эпителиально-мезенхимального перехода (EMT) и внеклеточного матрикса (ECM) были менее подавлены в адипогенных клетках после перепрограммирования (положительные коэффициенты взаимодействия Adipogenic: Reprogramming), тогда как наборы гликолитических генов были менее активированы (отрицательные коэффициенты взаимодействия). (F) Кластеризация набора генов со значимым типом клеток: взаимодействия при перепрограммировании выявляют заметные различия в величине эффекта перепрограммирования для разных типов клеток. Напротив, величина эффекта в зависимости от возраста в пределах одного и того же типа клеток довольно согласована.
Рисунок S6: Идентичность клетки определяет эффекты временного перепрограммирования.(A) Кратковременно перепрограммированные адипогенные клетки и МСК из наших полицистронных экспериментов были встроены в совместное латентное пространство с scVI, чтобы обеспечить прямое сравнение размеров эффекта. (B) ANOVA показал, что взаимодействия типа клетки: перепрограммирование составляют примерно 11% от общей неостаточной вариации, объясняемой ковариатами. (C) Мы повторно вычислили величины старения по максимальному среднему расхождению для контрольных адипогенных клеток и МСК в суставном латентном пространстве, чтобы учесть прямое сравнение. Мы обнаружили, что степень старения была значительно выше в адипогенных клетках (p 0,01, критерий суммы рангов Вилкоксона). (D) Мы выполнили дифференциальную экспрессию, чтобы идентифицировать гены со значительными взаимодействиями типа клетки: перепрограммирование. Качественный анализ экспрессии генов в латентном пространстве выявляет изменения эффектов, специфичных для типов клеток, по траекториям репрограммирования. (E) Мы использовали анализ генной онтологии, чтобы исследовать наиболее значимые взаимодействия типа клетки: перепрограммирование. Наборы генов эпителиально-мезенхимального перехода (EMT) и внеклеточного матрикса (ECM) были менее подавлены в адипогенных клетках после перепрограммирования (положительные коэффициенты взаимодействия Adipogenic: Reprogramming), тогда как наборы гликолитических генов были менее активированы (отрицательные коэффициенты взаимодействия). (F) Кластеризация набора генов со значимым типом клеток: взаимодействия при перепрограммировании выявляют заметные различия в величине эффекта перепрограммирования для разных типов клеток. Напротив, величина эффекта в зависимости от возраста в пределах одного и того же типа клеток довольно согласована.
 Рисунок S7: гены плюрипотентности активируются в подмножестве клеток в течение 2-4 дней после экспрессии OSKM в эмбриональных фибробластах мыши (MEF).(A) UMAP-проекция профилей мРНК отдельных клеток из MEF, подвергающихся перепрограммированию с использованием индуцируемого тетрациклином аллеля OSKM зародышевой линии [27]. Перепрограммирование вызывает новые состояния клеток всего через 2 дня, что согласуется с сильными эффектами, которые мы наблюдаем после короткого импульса OSKM. (B) Экспрессия маркеров плюрипотентности (Nanog, Rragd) и генов ответа на раннее репрограммирование (другие) в течение периода времени перепрограммирования MEF. Мы наблюдаем индукцию генов ответа на раннее перепрограммирование в соответствии с нашими данными временного перепрограммирования. Мы также наблюдаем активацию регулятора плюрипотентности Nanog в начале временного курса (день 2, 4), что согласуется с нашими временными результатами перепрограммирования. (C) Экспрессия плюрипотентности и ранних генов перепрограммирования через временной интервал перепрограммирования MEF. Nanog значительно усиливается только через 2 дня перепрограммирования (q 0,1, оценка логарифмического изменения Монте-Карло).
Рисунок S7: гены плюрипотентности активируются в подмножестве клеток в течение 2-4 дней после экспрессии OSKM в эмбриональных фибробластах мыши (MEF).(A) UMAP-проекция профилей мРНК отдельных клеток из MEF, подвергающихся перепрограммированию с использованием индуцируемого тетрациклином аллеля OSKM зародышевой линии [27]. Перепрограммирование вызывает новые состояния клеток всего через 2 дня, что согласуется с сильными эффектами, которые мы наблюдаем после короткого импульса OSKM. (B) Экспрессия маркеров плюрипотентности (Nanog, Rragd) и генов ответа на раннее репрограммирование (другие) в течение периода времени перепрограммирования MEF. Мы наблюдаем индукцию генов ответа на раннее перепрограммирование в соответствии с нашими данными временного перепрограммирования. Мы также наблюдаем активацию регулятора плюрипотентности Nanog в начале временного курса (день 2, 4), что согласуется с нашими временными результатами перепрограммирования. (C) Экспрессия плюрипотентности и ранних генов перепрограммирования через временной интервал перепрограммирования MEF. Nanog значительно усиливается только через 2 дня перепрограммирования (q 0,1, оценка логарифмического изменения Монте-Карло).
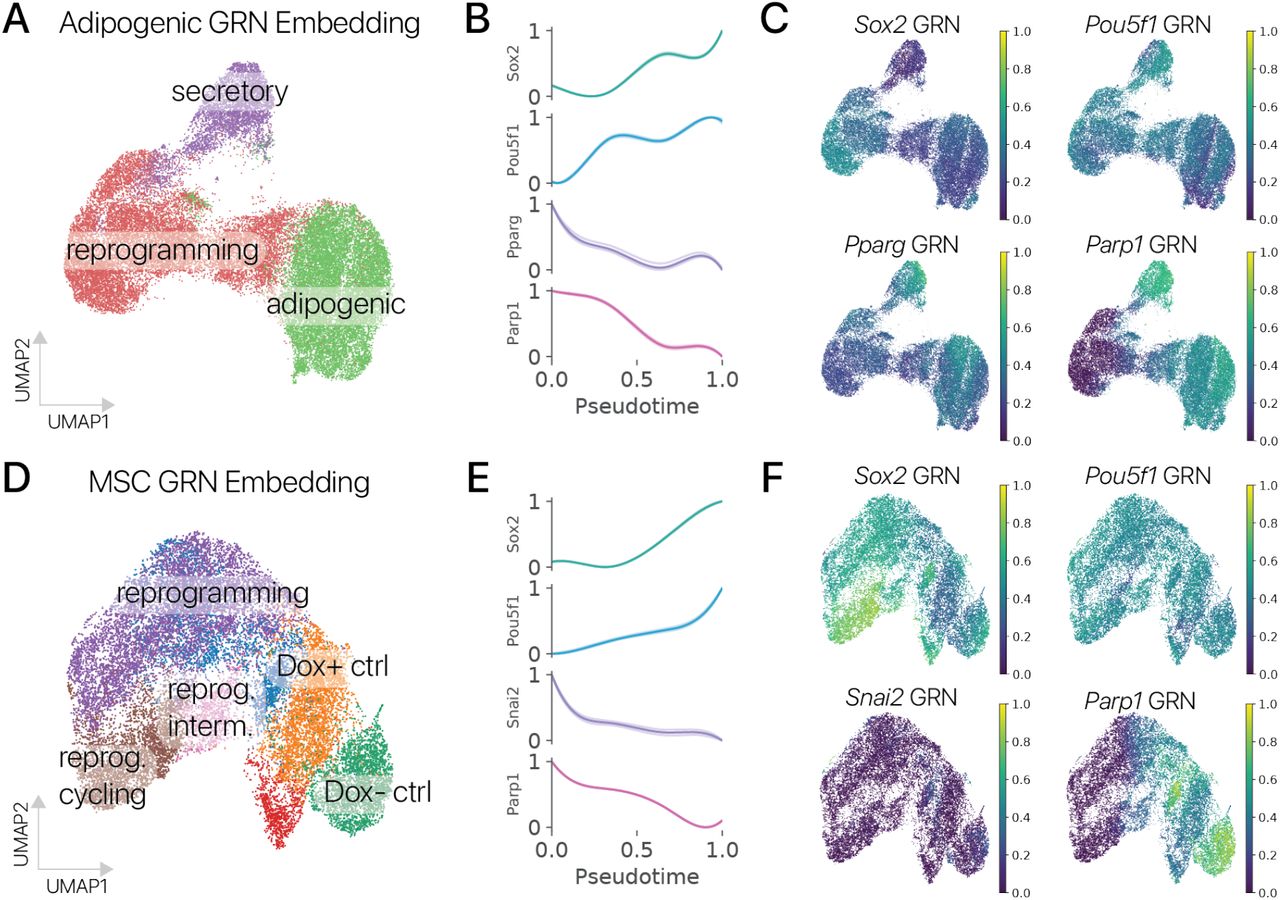 Рисунок S8: Анализ генной регуляторной сети (GRN) показывает подавление GRN идентичности соматических клеток во время временного репрограммирования.Мы извлекли направленный граф GRN из базы данных TRRUST, связывая факторы транскрипции мышей с прямыми генами-мишенями [30]. Затем мы оценили активность GRN в временно перепрограммированных адипогенных клетках и МСК, используя подход интеграции на основе рангов, заимствованный из SCENIC [29]. (A) Адипогенные клетки, встроенные в транскрипционное пространство, полученное из оценок активности GRN. Показатели GRN точно отражают эффекты перепрограммирования и различия состояний клеток в контрольных популяциях. (B) баллы GRN в псевдовремя перепрограммирования (чем выше, тем больше перепрограммировано). Мы обнаружили, что GRN плюрипотентности (Sox2, Pou5f1) увеличиваются, как и ожидалось, при перепрограммировании, в то время как GRN с адипогенной идентичностью (Pparg, Parp1) подавляются (тест Вальда, биномиальный GLM, p 0,01). (C) GRN плюрипотентности (вверху) и GRN с адипогенной идентичностью (внизу), проецируемые в латентное пространство оценки GRN, четко различают контрольные и временно перепрограммированные клетки. (D) MSC, полученные из мышц, встроенные с использованием показателей активности GRN. Показатели GRN снова отражают изменения состояния клеток, вызванные временным перепрограммированием. (E) GRN плюрипотентности увеличиваются, как и ожидалось, с перепрограммированием в MSC, в то время как GRN мезенхимальной идентичности (Snai2, Parp1) подавляются (тест Вальда, биномиальный GLM, p 0,01). (F) Плюрипотентность (вверху) и мезенхимальная идентичность (внизу) оценки GRN в латентном пространстве GRN показывают явное обогащение для перепрограммированной и контрольной популяций соответственно.
Рисунок S8: Анализ генной регуляторной сети (GRN) показывает подавление GRN идентичности соматических клеток во время временного репрограммирования.Мы извлекли направленный граф GRN из базы данных TRRUST, связывая факторы транскрипции мышей с прямыми генами-мишенями [30]. Затем мы оценили активность GRN в временно перепрограммированных адипогенных клетках и МСК, используя подход интеграции на основе рангов, заимствованный из SCENIC [29]. (A) Адипогенные клетки, встроенные в транскрипционное пространство, полученное из оценок активности GRN. Показатели GRN точно отражают эффекты перепрограммирования и различия состояний клеток в контрольных популяциях. (B) баллы GRN в псевдовремя перепрограммирования (чем выше, тем больше перепрограммировано). Мы обнаружили, что GRN плюрипотентности (Sox2, Pou5f1) увеличиваются, как и ожидалось, при перепрограммировании, в то время как GRN с адипогенной идентичностью (Pparg, Parp1) подавляются (тест Вальда, биномиальный GLM, p 0,01). (C) GRN плюрипотентности (вверху) и GRN с адипогенной идентичностью (внизу), проецируемые в латентное пространство оценки GRN, четко различают контрольные и временно перепрограммированные клетки. (D) MSC, полученные из мышц, встроенные с использованием показателей активности GRN. Показатели GRN снова отражают изменения состояния клеток, вызванные временным перепрограммированием. (E) GRN плюрипотентности увеличиваются, как и ожидалось, с перепрограммированием в MSC, в то время как GRN мезенхимальной идентичности (Snai2, Parp1) подавляются (тест Вальда, биномиальный GLM, p 0,01). (F) Плюрипотентность (вверху) и мезенхимальная идентичность (внизу) оценки GRN в латентном пространстве GRN показывают явное обогащение для перепрограммированной и контрольной популяций соответственно.
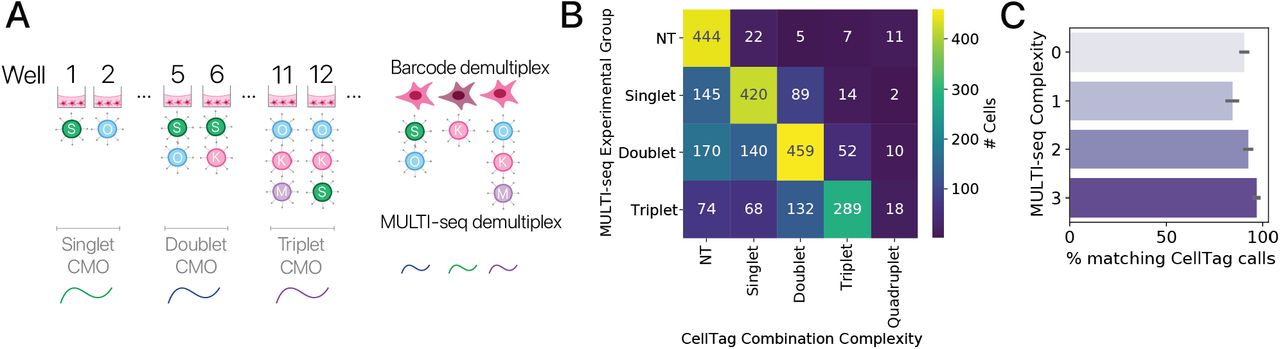 Рисунок S9: Маркировка MULTI-seq комбинаторной сложности и демультиплексирование на основе выраженного штрих-кода демонстрируют строгое соответствие(А) Схема эксперимента. Мы выполнили пилотный объединенный скрининг в формате массива и обозначили количество уникальных факторов в каждом состоянии лечения с помощью MULTI-seq. Затем мы сравнили метки, полученные с помощью MULTI-seq, с нашей системой выраженного штрих-кода (CellTag) для демультиплексирования комбинаторных возмущений. Присвоение меток MULTI-seq несовершенно, и выраженные штрих-коды могут не обнаруживать ошибок, поэтому мы не ожидаем идеального соответствия 1: 1. (B) Тепловая карта соответствия между метками сложности, полученными из MULTI-seq, и выраженными штрих-кодами для ячеек с достоверной меткой, полученной из MULTI-seq. Мы обнаружили, что ожидаемая сложность, основанная на штрих-кодах MULTI-seq, является доминирующим режимом для выраженных классификаций штрих-кодов. Мы ожидаем, что в нижнем треугольнике матрицы появятся элементы из-за (1) неудачных обнаружений штрих-кода или (2) ячеек, которые получили меньше целевого числа уникальных факторов в данном состоянии из-за стохастичности преобразования. Поэтому мы считаем все метки в нижней треугольной матрице точными. Очень мало ячеек выходит за пределы нижнего треугольника, вероятно, из-за неправильных вызовов штрих-кода MULTI-seq. (C) Соответствие меток, количественно оцененных по меткам условий лечения MULTI-seq (среднее + 95% ДИ). Все сложности показывают высокую степень соответствия (80% совпадений calls).
Рисунок S9: Маркировка MULTI-seq комбинаторной сложности и демультиплексирование на основе выраженного штрих-кода демонстрируют строгое соответствие(А) Схема эксперимента. Мы выполнили пилотный объединенный скрининг в формате массива и обозначили количество уникальных факторов в каждом состоянии лечения с помощью MULTI-seq. Затем мы сравнили метки, полученные с помощью MULTI-seq, с нашей системой выраженного штрих-кода (CellTag) для демультиплексирования комбинаторных возмущений. Присвоение меток MULTI-seq несовершенно, и выраженные штрих-коды могут не обнаруживать ошибок, поэтому мы не ожидаем идеального соответствия 1: 1. (B) Тепловая карта соответствия между метками сложности, полученными из MULTI-seq, и выраженными штрих-кодами для ячеек с достоверной меткой, полученной из MULTI-seq. Мы обнаружили, что ожидаемая сложность, основанная на штрих-кодах MULTI-seq, является доминирующим режимом для выраженных классификаций штрих-кодов. Мы ожидаем, что в нижнем треугольнике матрицы появятся элементы из-за (1) неудачных обнаружений штрих-кода или (2) ячеек, которые получили меньше целевого числа уникальных факторов в данном состоянии из-за стохастичности преобразования. Поэтому мы считаем все метки в нижней треугольной матрице точными. Очень мало ячеек выходит за пределы нижнего треугольника, вероятно, из-за неправильных вызовов штрих-кода MULTI-seq. (C) Соответствие меток, количественно оцененных по меткам условий лечения MULTI-seq (среднее + 95% ДИ). Все сложности показывают высокую степень соответствия (80% совпадений calls).
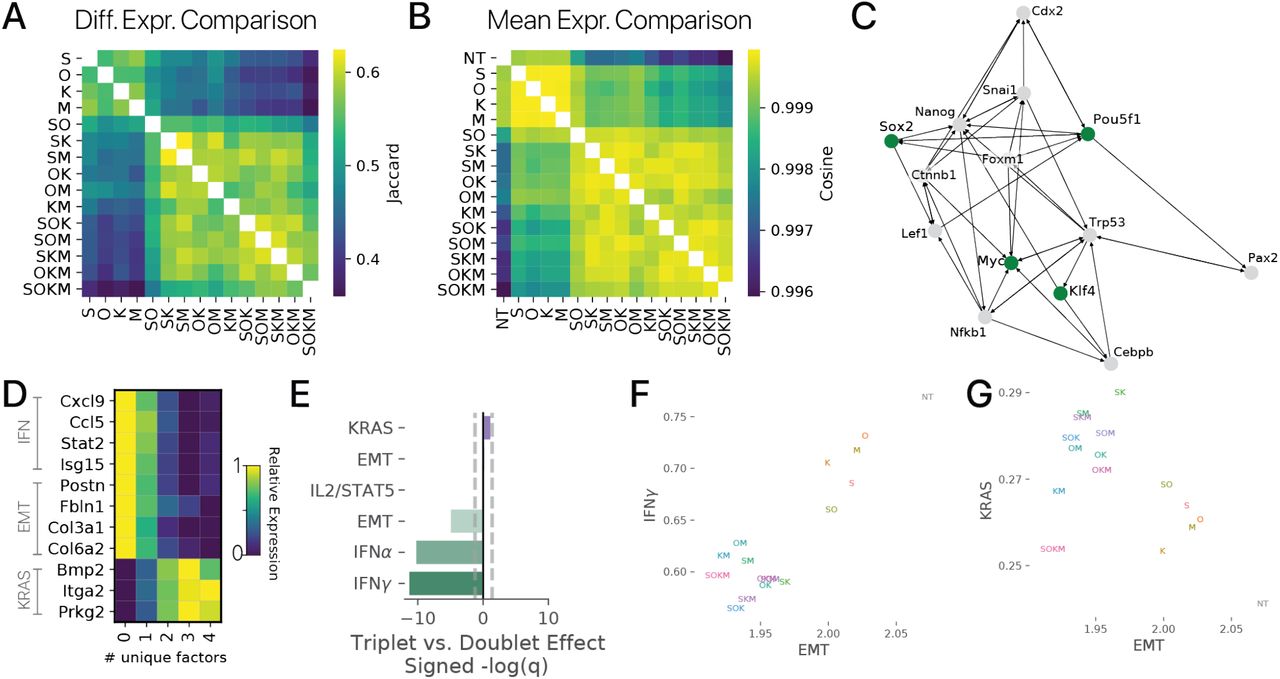 Рисунок S10: Комбинации факторов Яманака более высокого порядка оказывают аналогичные транскрипционные эффекты.(A) Сравнение наборов дифференциально экспрессируемых генов (DEG) относительно контроля для каждого возмущения. Индекс Жаккара измеряет количество общих генов в двух наборах как долю от общего числа уникальных генов в обоих наборах. Подобно результатам, полученным с помощью нашего классификатора возмущений, мы обнаруживаем, что комбинации факторов Яманака с аналогичной сложностью вызывают аналогичные транскрипционные эффекты после кратковременного импульса. (B) Сравнение средних векторов экспрессии генов для каждой комбинации фактора Яманака (NT: необработанные контроли) с использованием косинусного сходства (положительные значения более похожи). Мы снова обнаружили, что комбинации более высокого порядка вызывают аналогичные транскрипционные эффекты. (C) Принудительно управляемая визуализация компоновки соединений в регуляторной сети гена плюрипотентности, извлеченной из базы данных TRRUST. Факторы Яманки выделены зеленым цветом. Все три подмножества факторов Яманака могут активировать четвертый фактор. (D) Сравнение интерферона (IFN), эпителиально-мезенхимального перехода (EMT) и сигнатур генов KRAS в клетках с различным количеством уникальных факторов перепрограммирования. Наибольшие различия между комбинациями трех и двух факторов заключаются в более сильном подавлении программ IFN и EMT и более сильной активации программы передачи сигналов KRAS. (E) Обогащенный анализ онтологии генов с наборами генов MSigDB Hallmark подтверждает результаты на уровне генов в (D). (F) Программная активность EMT и IFNγ тесно коррелирована (r 0,9), что позволяет предположить, что они подавляются общим механизмом. (G) Программные мероприятия EMT и KRAS антикоррелированы (r - 0,6), что позволяет предположить, что эти эффекты также могут иметь общий механизм во время процесса перепрограммирования.
Рисунок S10: Комбинации факторов Яманака более высокого порядка оказывают аналогичные транскрипционные эффекты.(A) Сравнение наборов дифференциально экспрессируемых генов (DEG) относительно контроля для каждого возмущения. Индекс Жаккара измеряет количество общих генов в двух наборах как долю от общего числа уникальных генов в обоих наборах. Подобно результатам, полученным с помощью нашего классификатора возмущений, мы обнаруживаем, что комбинации факторов Яманака с аналогичной сложностью вызывают аналогичные транскрипционные эффекты после кратковременного импульса. (B) Сравнение средних векторов экспрессии генов для каждой комбинации фактора Яманака (NT: необработанные контроли) с использованием косинусного сходства (положительные значения более похожи). Мы снова обнаружили, что комбинации более высокого порядка вызывают аналогичные транскрипционные эффекты. (C) Принудительно управляемая визуализация компоновки соединений в регуляторной сети гена плюрипотентности, извлеченной из базы данных TRRUST. Факторы Яманки выделены зеленым цветом. Все три подмножества факторов Яманака могут активировать четвертый фактор. (D) Сравнение интерферона (IFN), эпителиально-мезенхимального перехода (EMT) и сигнатур генов KRAS в клетках с различным количеством уникальных факторов перепрограммирования. Наибольшие различия между комбинациями трех и двух факторов заключаются в более сильном подавлении программ IFN и EMT и более сильной активации программы передачи сигналов KRAS. (E) Обогащенный анализ онтологии генов с наборами генов MSigDB Hallmark подтверждает результаты на уровне генов в (D). (F) Программная активность EMT и IFNγ тесно коррелирована (r 0,9), что позволяет предположить, что они подавляются общим механизмом. (G) Программные мероприятия EMT и KRAS антикоррелированы (r - 0,6), что позволяет предположить, что эти эффекты также могут иметь общий механизм во время процесса перепрограммирования.
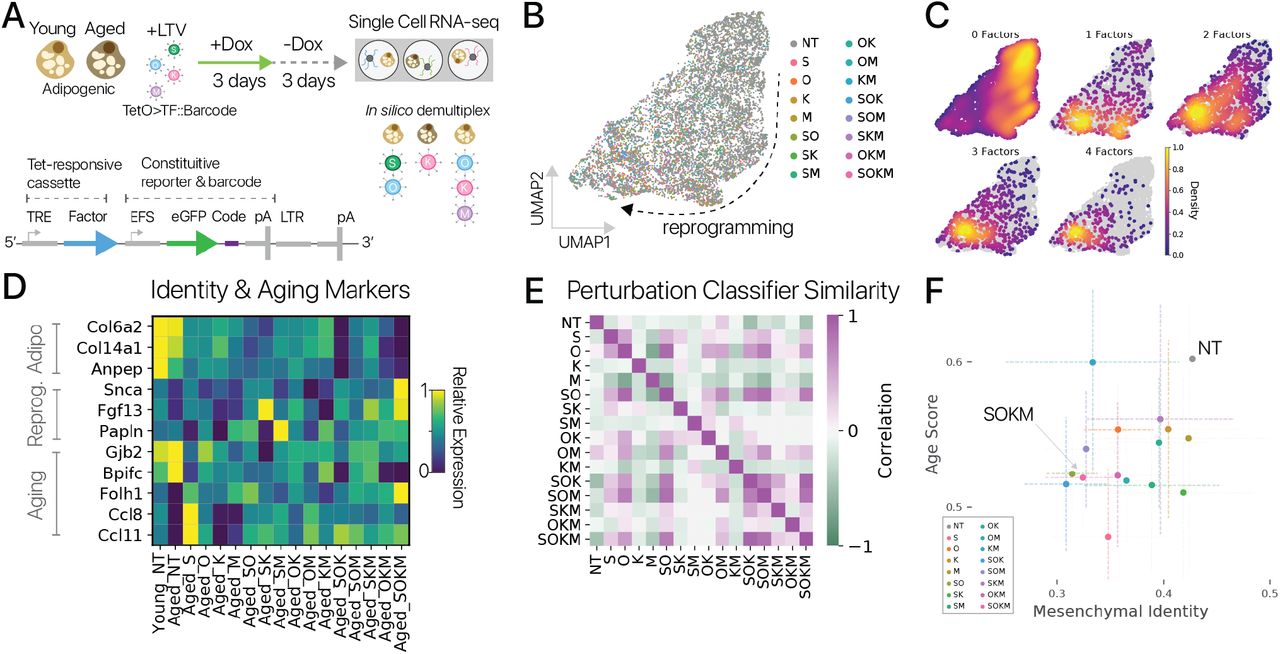 Рисунок S11: Объединенный скрининг адипогенных клеток дает результаты, аналогичные скринингу на МСК.(A) Схема объединенных скрининговых экспериментов с фактором Яманака. Молодые и старые адипогенные клетки трансдуцировали лентивирусами, каждый из которых нес один индуцибельный фактор Яманака с выраженными штрих-кодами (внизу). Перепрограммирование индуцировали для 3-дневного пульса / 3-дневного преследования (n = 5 животных на возраст в двух независимых экспериментах). Клетки были профилированы с помощью одиночной клеточной РНК-seq, и уникальные комбинации факторов Яманака были демультиплексированы in silico на основе выраженных штрих-кодов. (B) Клетки из объединенного экрана, встроенные с использованием scNym, спроецированные с помощью UMAP и помеченные обнаруженными факторами перепрограммирования (9000+ клеток). (C) Плотность клеток, нарушенных различным количеством факторов перепрограммирования при внедрении UMAP. Комбинации более высокого порядка показывают больший сдвиг транскрипции по сравнению с контрольными клетками. (D) Адипогенные маркерные гены (вверху) уменьшаются, а маркерные гены репрограммирования (в центре) увеличиваются по мере увеличения комбинаторной сложности (количества уникальных факторов). Гены старения (нижний) также кажутся ближе к юношескому уровню с более сложными нарушениями. (E) Матрица сходства между различными комбинациями факторов Яманака, извлеченными из модели классификации возмущений клеток. Сходство классификатора менее драматично, чем в МСК, но наиболее сильное сходство снова наблюдается для комбинаций факторов Яманака более высокого порядка. (F) Показатели идентичности мезенхимальных клеток, полученные на основе моделей scNym, и оценки набора генов старения в старых клетках, перепрограммированных с помощью различных комбинаций факторов (средние значения ± 95% ДИ). Многие комбинации факторов Яманака значительно снижали как показатель идентичности мезенхимальных клеток, так и показатель возраста по сравнению со старыми контрольными клетками (NT) (тесты Вальда, p 0,05). Однако возрастные баллы и баллы идентичности не были хорошо коррелированы, предполагая, что подавление идентичности клеток и омоложение не тесно связаны (р = 0,19, р 0,48, Спирмен).
Рисунок S11: Объединенный скрининг адипогенных клеток дает результаты, аналогичные скринингу на МСК.(A) Схема объединенных скрининговых экспериментов с фактором Яманака. Молодые и старые адипогенные клетки трансдуцировали лентивирусами, каждый из которых нес один индуцибельный фактор Яманака с выраженными штрих-кодами (внизу). Перепрограммирование индуцировали для 3-дневного пульса / 3-дневного преследования (n = 5 животных на возраст в двух независимых экспериментах). Клетки были профилированы с помощью одиночной клеточной РНК-seq, и уникальные комбинации факторов Яманака были демультиплексированы in silico на основе выраженных штрих-кодов. (B) Клетки из объединенного экрана, встроенные с использованием scNym, спроецированные с помощью UMAP и помеченные обнаруженными факторами перепрограммирования (9000+ клеток). (C) Плотность клеток, нарушенных различным количеством факторов перепрограммирования при внедрении UMAP. Комбинации более высокого порядка показывают больший сдвиг транскрипции по сравнению с контрольными клетками. (D) Адипогенные маркерные гены (вверху) уменьшаются, а маркерные гены репрограммирования (в центре) увеличиваются по мере увеличения комбинаторной сложности (количества уникальных факторов). Гены старения (нижний) также кажутся ближе к юношескому уровню с более сложными нарушениями. (E) Матрица сходства между различными комбинациями факторов Яманака, извлеченными из модели классификации возмущений клеток. Сходство классификатора менее драматично, чем в МСК, но наиболее сильное сходство снова наблюдается для комбинаций факторов Яманака более высокого порядка. (F) Показатели идентичности мезенхимальных клеток, полученные на основе моделей scNym, и оценки набора генов старения в старых клетках, перепрограммированных с помощью различных комбинаций факторов (средние значения ± 95% ДИ). Многие комбинации факторов Яманака значительно снижали как показатель идентичности мезенхимальных клеток, так и показатель возраста по сравнению со старыми контрольными клетками (NT) (тесты Вальда, p 0,05). Однако возрастные баллы и баллы идентичности не были хорошо коррелированы, предполагая, что подавление идентичности клеток и омоложение не тесно связаны (р = 0,19, р 0,48, Спирмен).
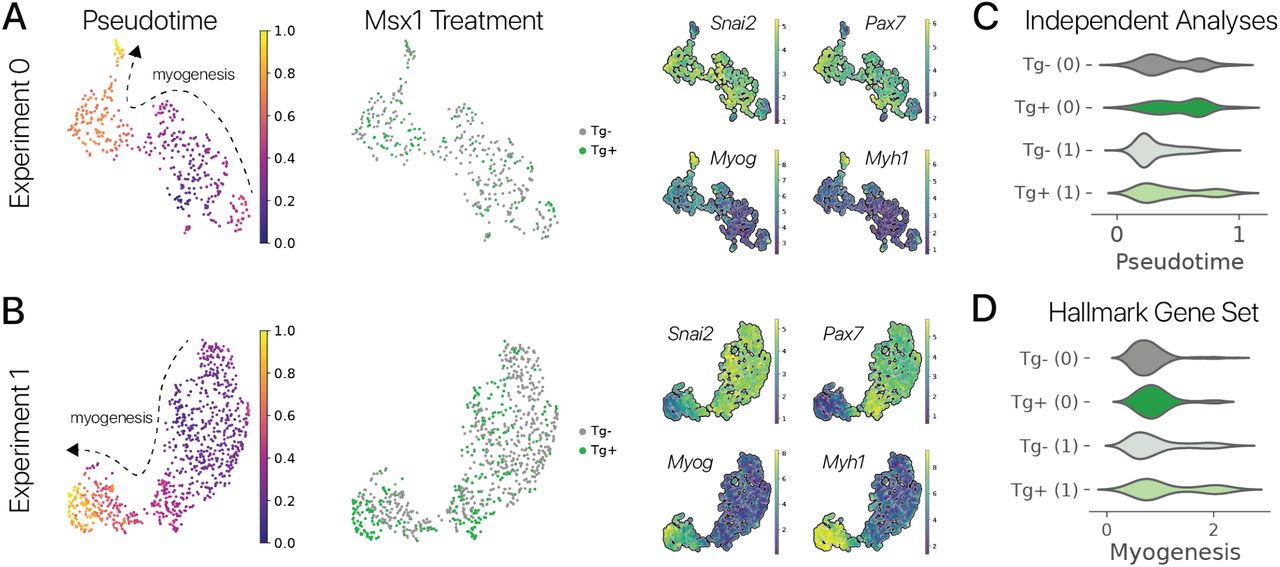 Рисунок S12: Импульсное перепрограммирование с фактором мультипотентности Msx1 увеличивает миогенез в старых миогенных клетках.Старые миогенные клетки обрабатывали кратковременным импульсом Msx1 в двух отдельных экспериментах. Эксперименты были проанализированы независимо ниже. (A) Псевдоанализ (слева) и анализ маркерного гена (справа) показывают траекторию миогенной дифференцировки как в первом (n = 2 животных, возраст 28 месяцев), так и (B) во втором независимом эксперименте (n = 3 животных, возраст 30 месяцев). ). (C) Мы выполнили анализ псевдовременности независимо в каждом экспериментальном состоянии. Как и в нашем интегрированном анализе (рис. 4), временно перепрограммированные клетки были значительно более дифференцированными, чем контрольные в обоих экспериментах (критерий суммы рангов Вилкоксона, p 0,01). (D) Мы оценили активность набора генов Hallmark Myogenesis (MSigDB). Оба эксперимента показали значительное увеличение активности набора миогенных генов после обработки временным перепрограммированием (критерий суммы рангов Вилкоксона, p 0,01).
Рисунок S12: Импульсное перепрограммирование с фактором мультипотентности Msx1 увеличивает миогенез в старых миогенных клетках.Старые миогенные клетки обрабатывали кратковременным импульсом Msx1 в двух отдельных экспериментах. Эксперименты были проанализированы независимо ниже. (A) Псевдоанализ (слева) и анализ маркерного гена (справа) показывают траекторию миогенной дифференцировки как в первом (n = 2 животных, возраст 28 месяцев), так и (B) во втором независимом эксперименте (n = 3 животных, возраст 30 месяцев). ). (C) Мы выполнили анализ псевдовременности независимо в каждом экспериментальном состоянии. Как и в нашем интегрированном анализе (рис. 4), временно перепрограммированные клетки были значительно более дифференцированными, чем контрольные в обоих экспериментах (критерий суммы рангов Вилкоксона, p 0,01). (D) Мы оценили активность набора генов Hallmark Myogenesis (MSigDB). Оба эксперимента показали значительное увеличение активности набора миогенных генов после обработки временным перепрограммированием (критерий суммы рангов Вилкоксона, p 0,01).
Дополнительные таблицы
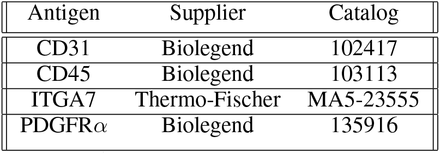
Опубликовано: 25 июня 2021 г.
Авторы: Csaba Kerepesi, Bohan Zhang, Sang-Goo Lee, Alexandre Trapp, Vadim N. Gladyshev
Оригинальная статья: Epigenetic clocks reveal a rejuvenation event during embryogenesis followed by aging
Перевод Ник Сестрин

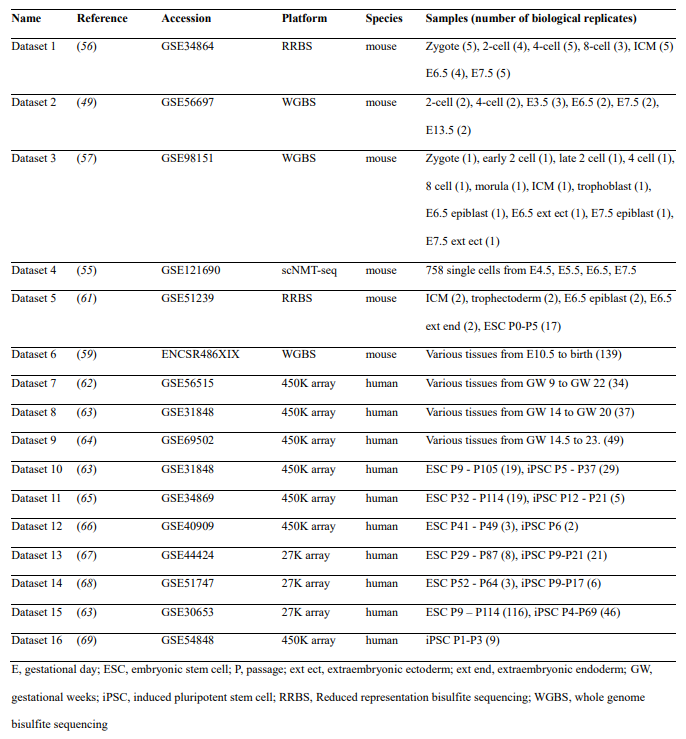
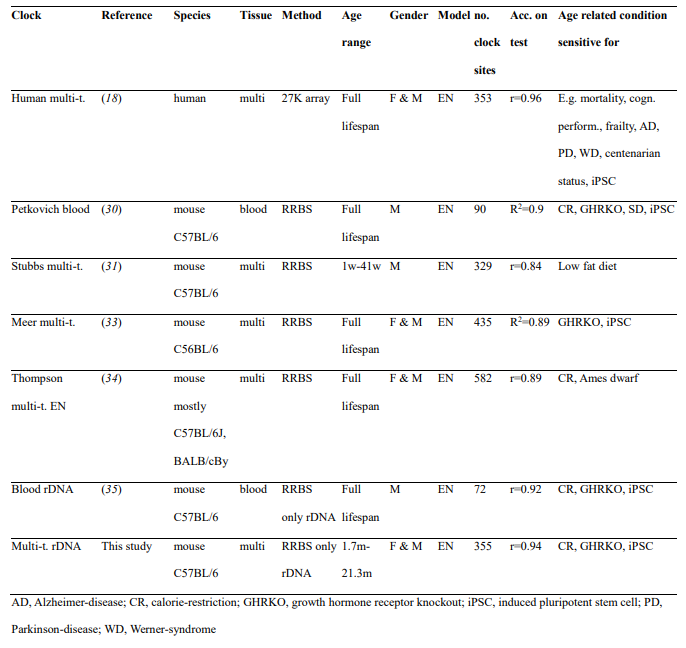
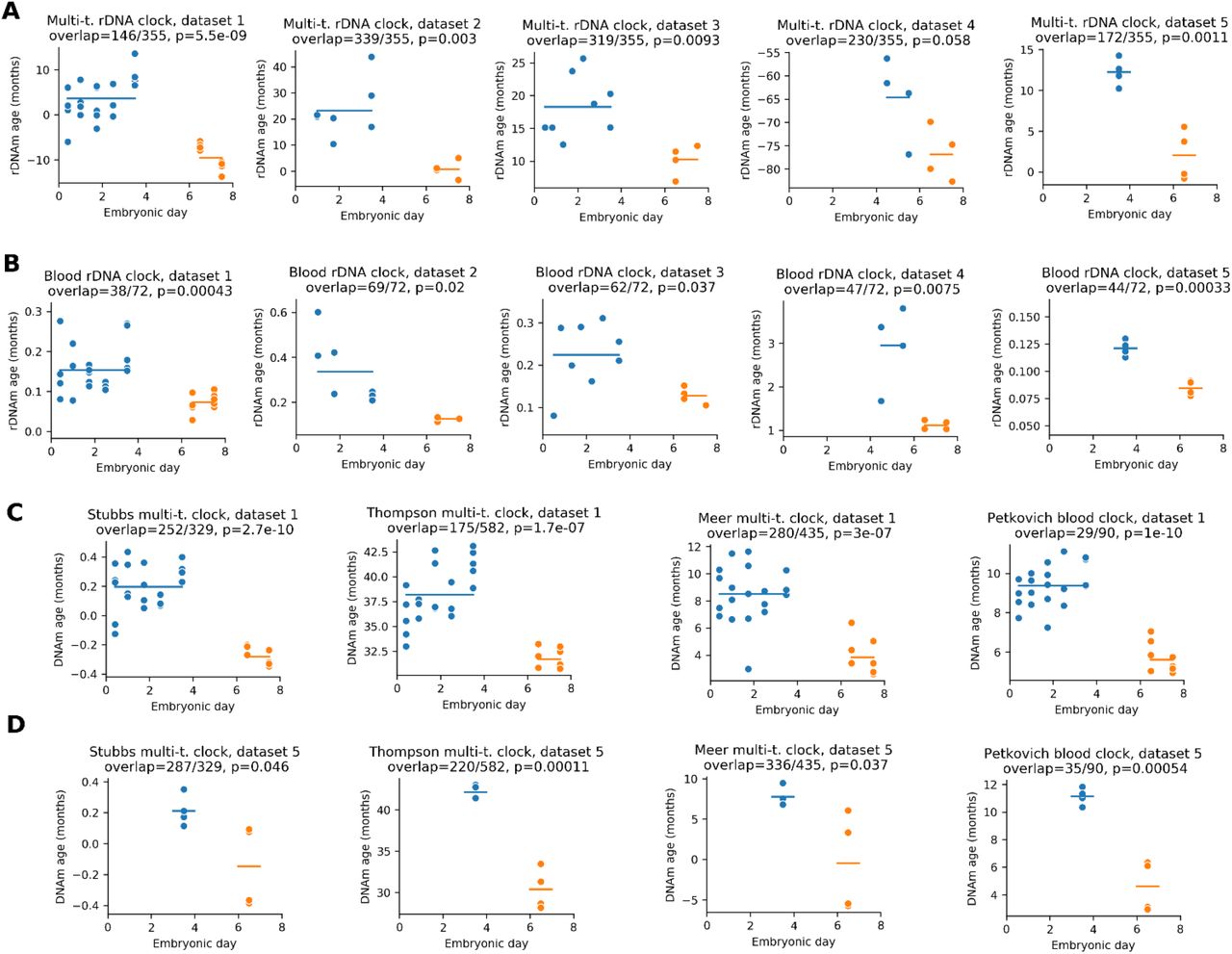
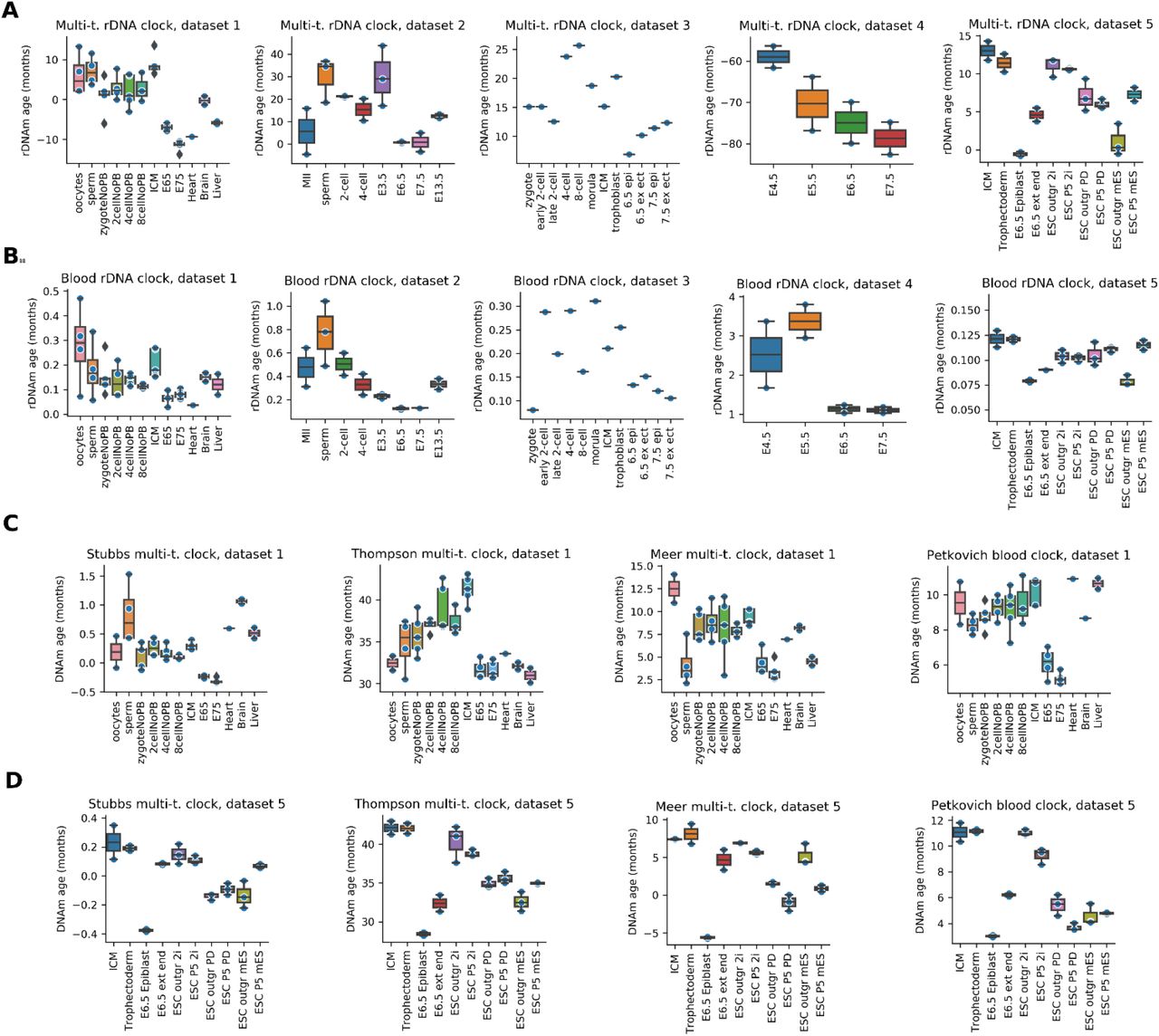
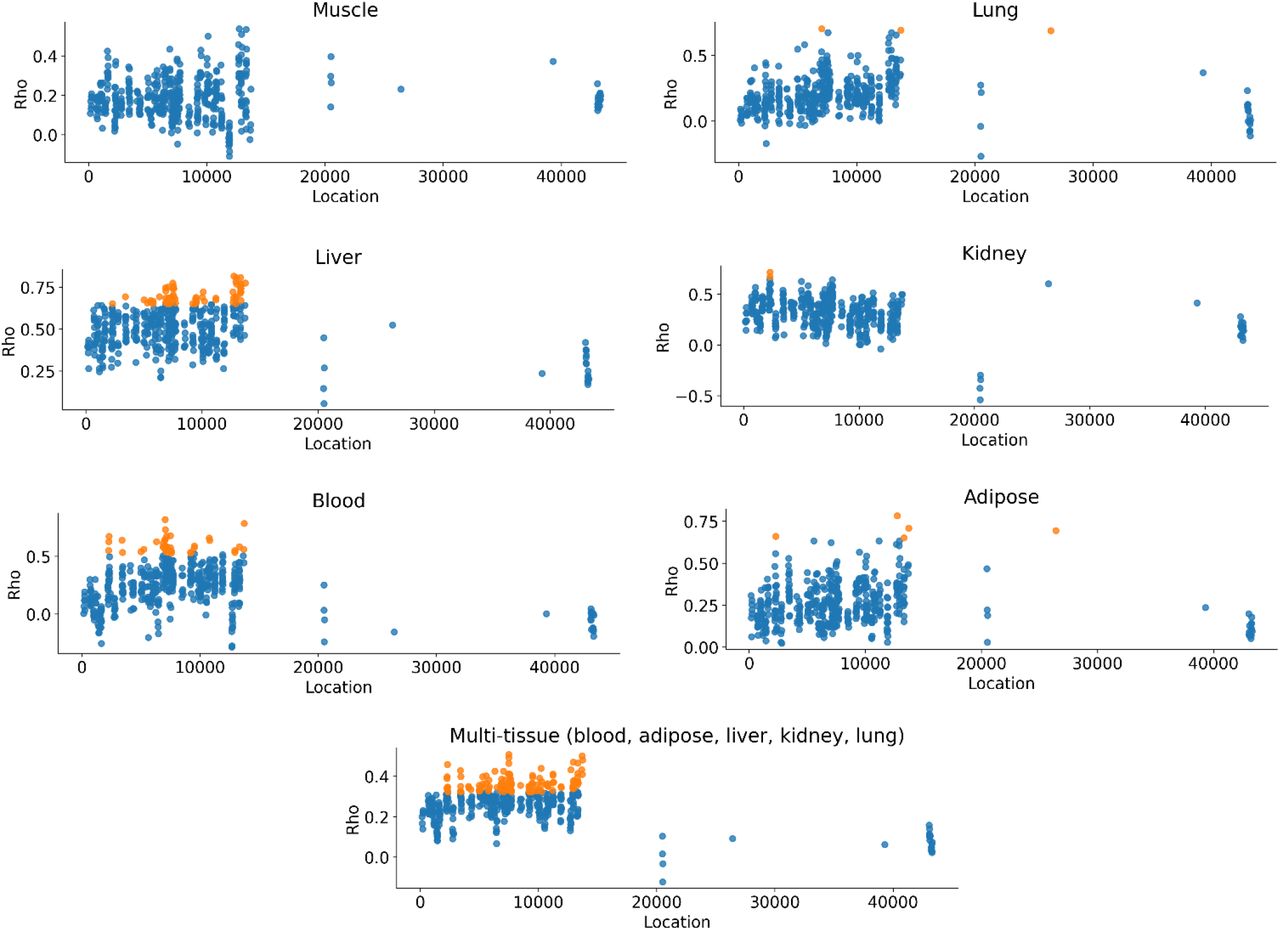
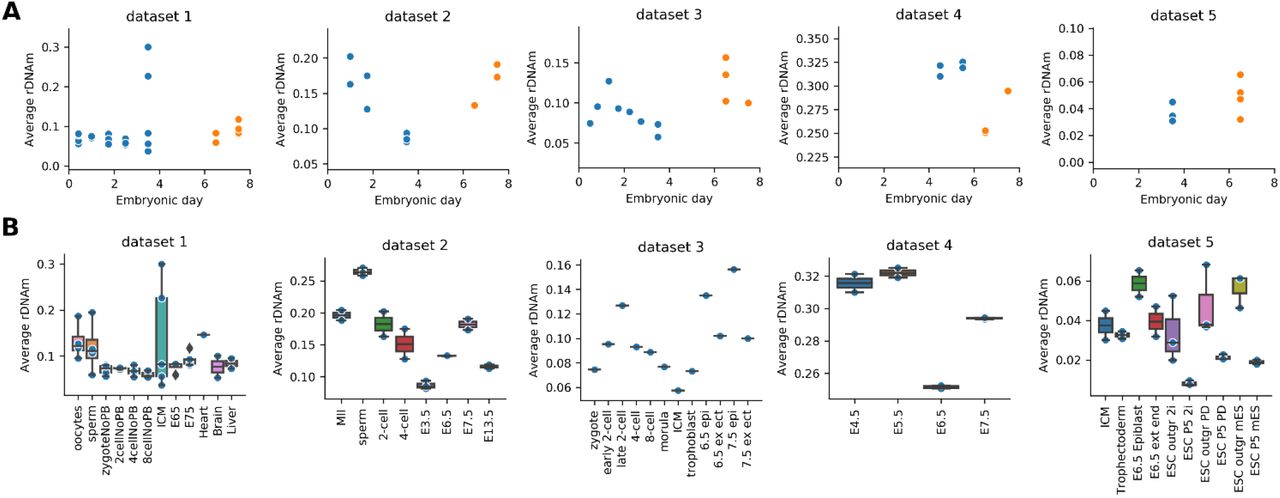
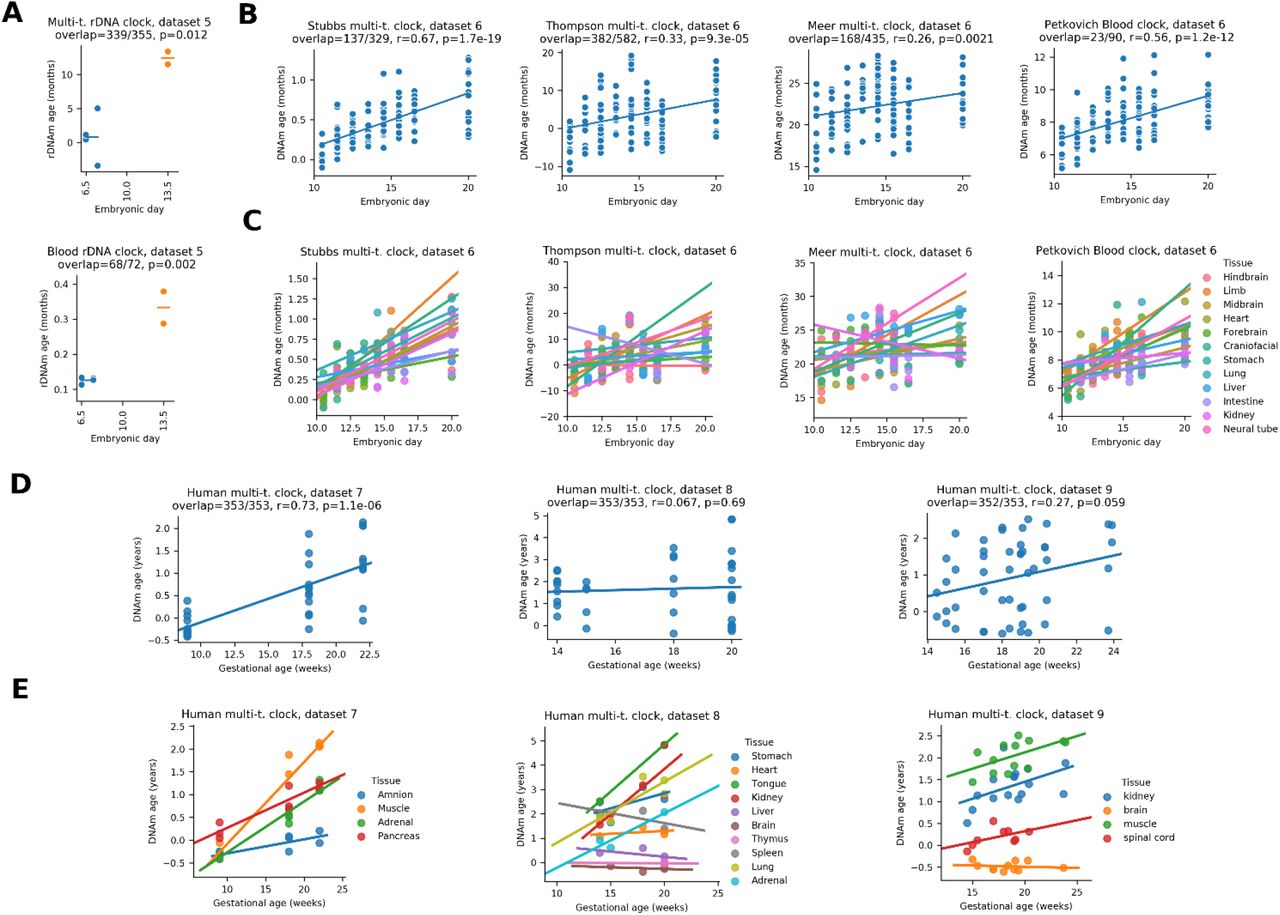
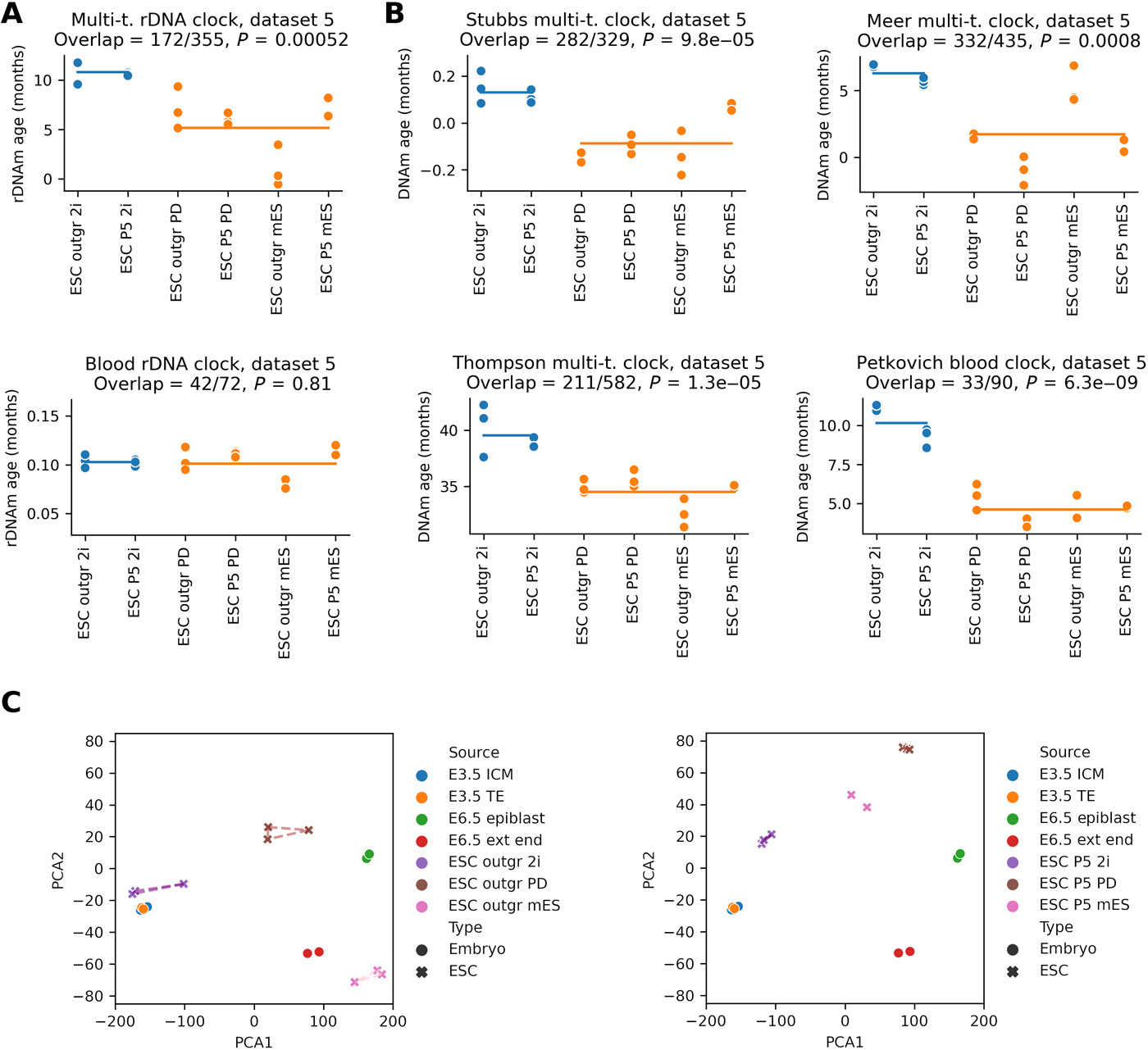
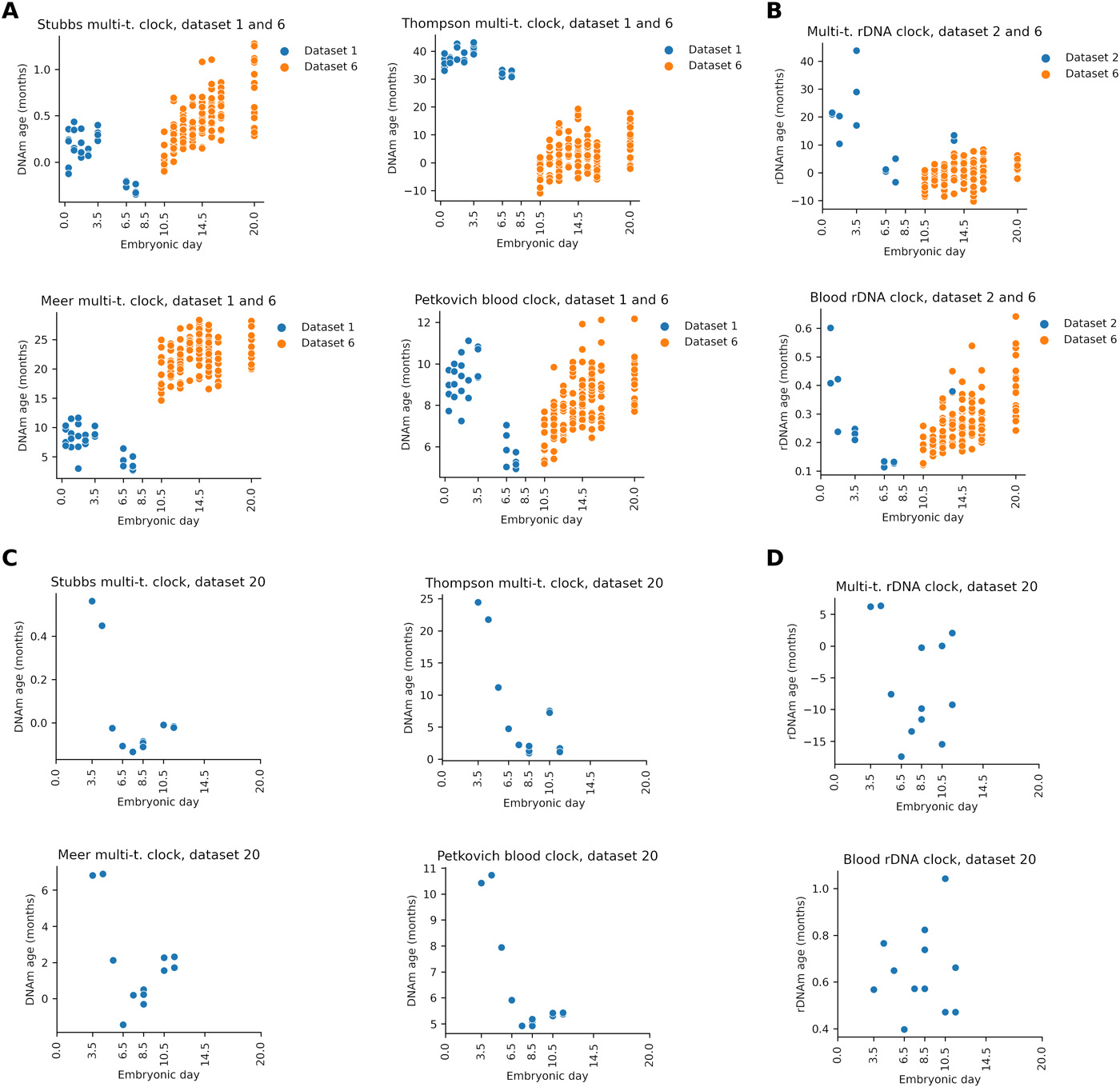
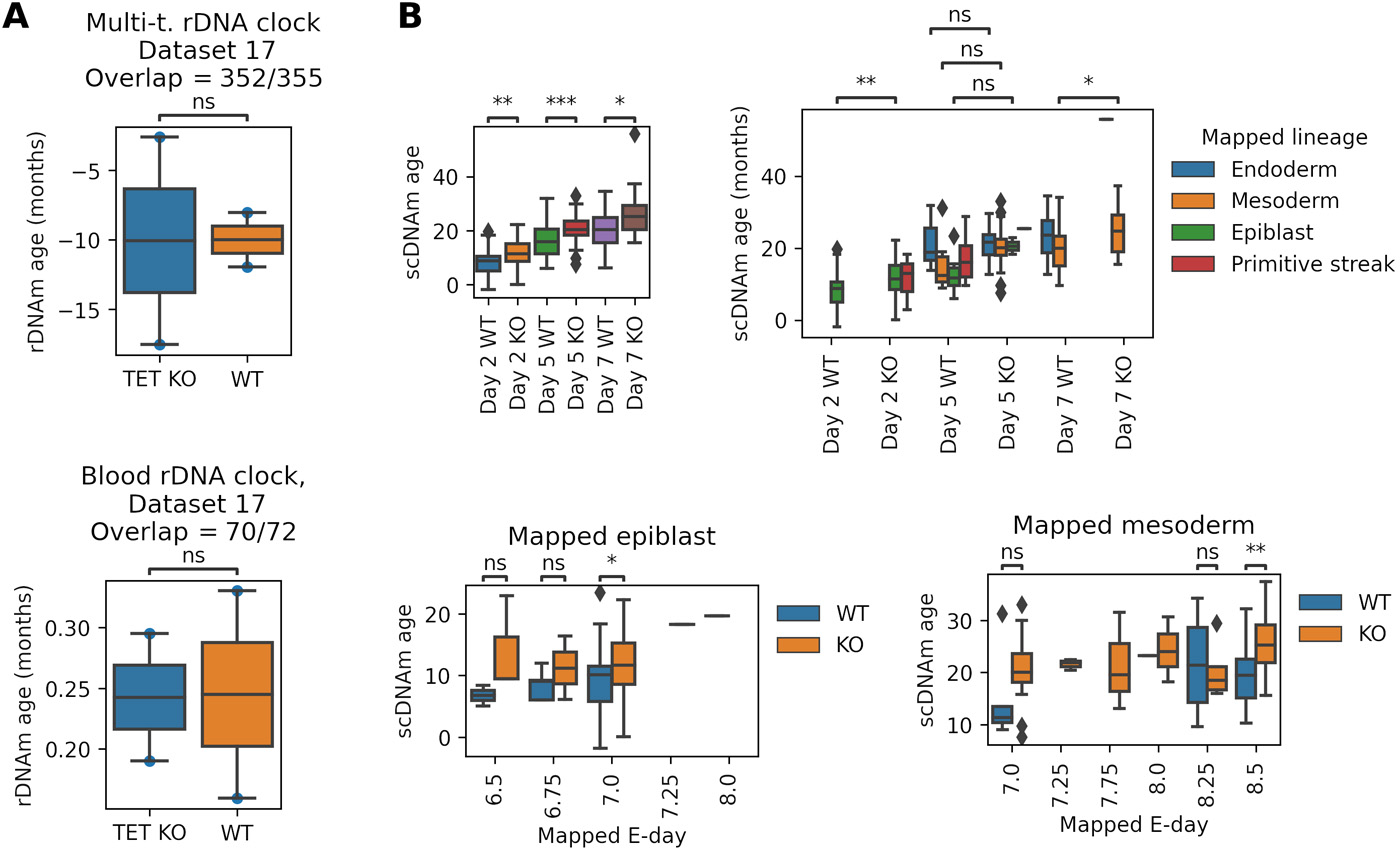
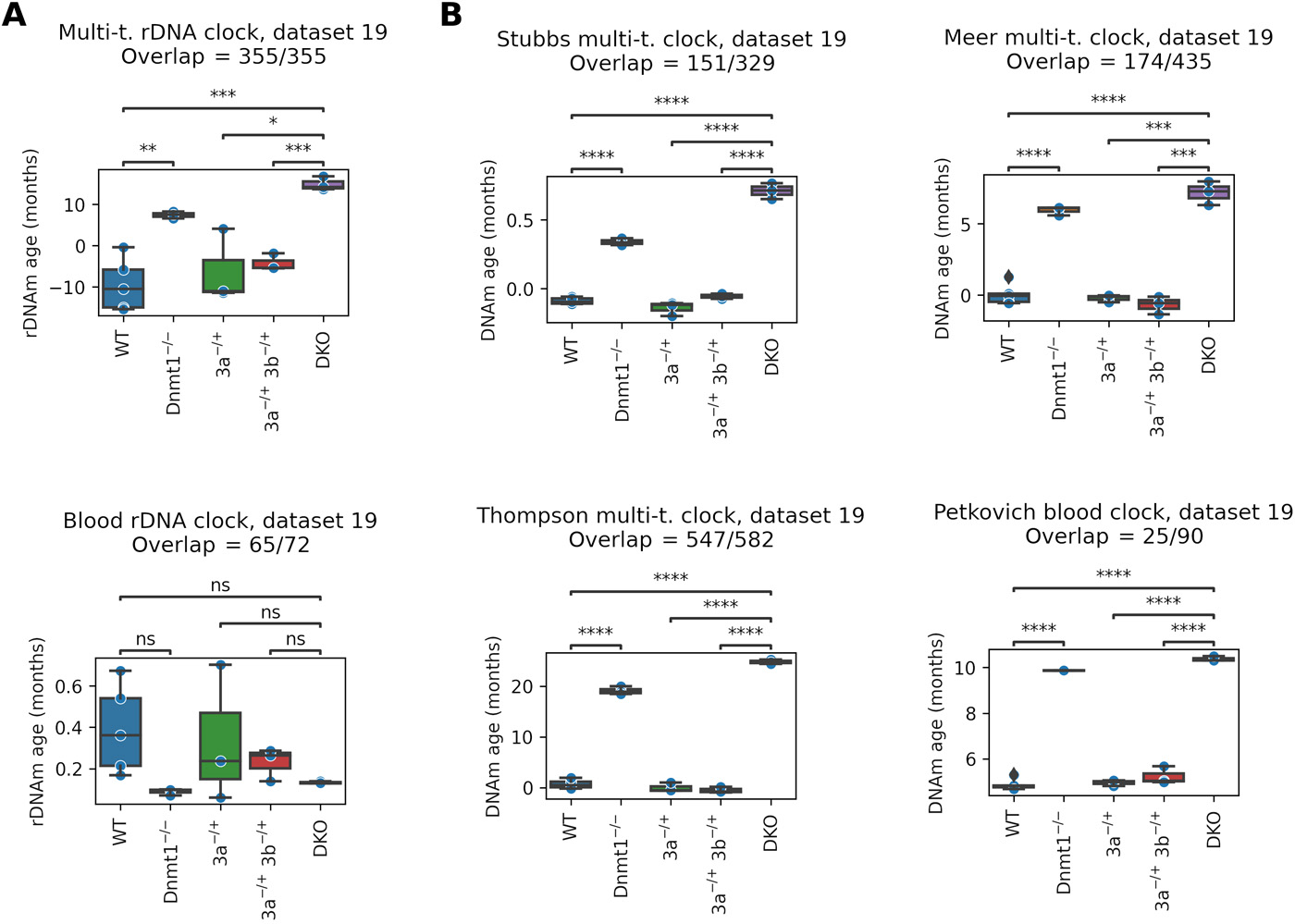
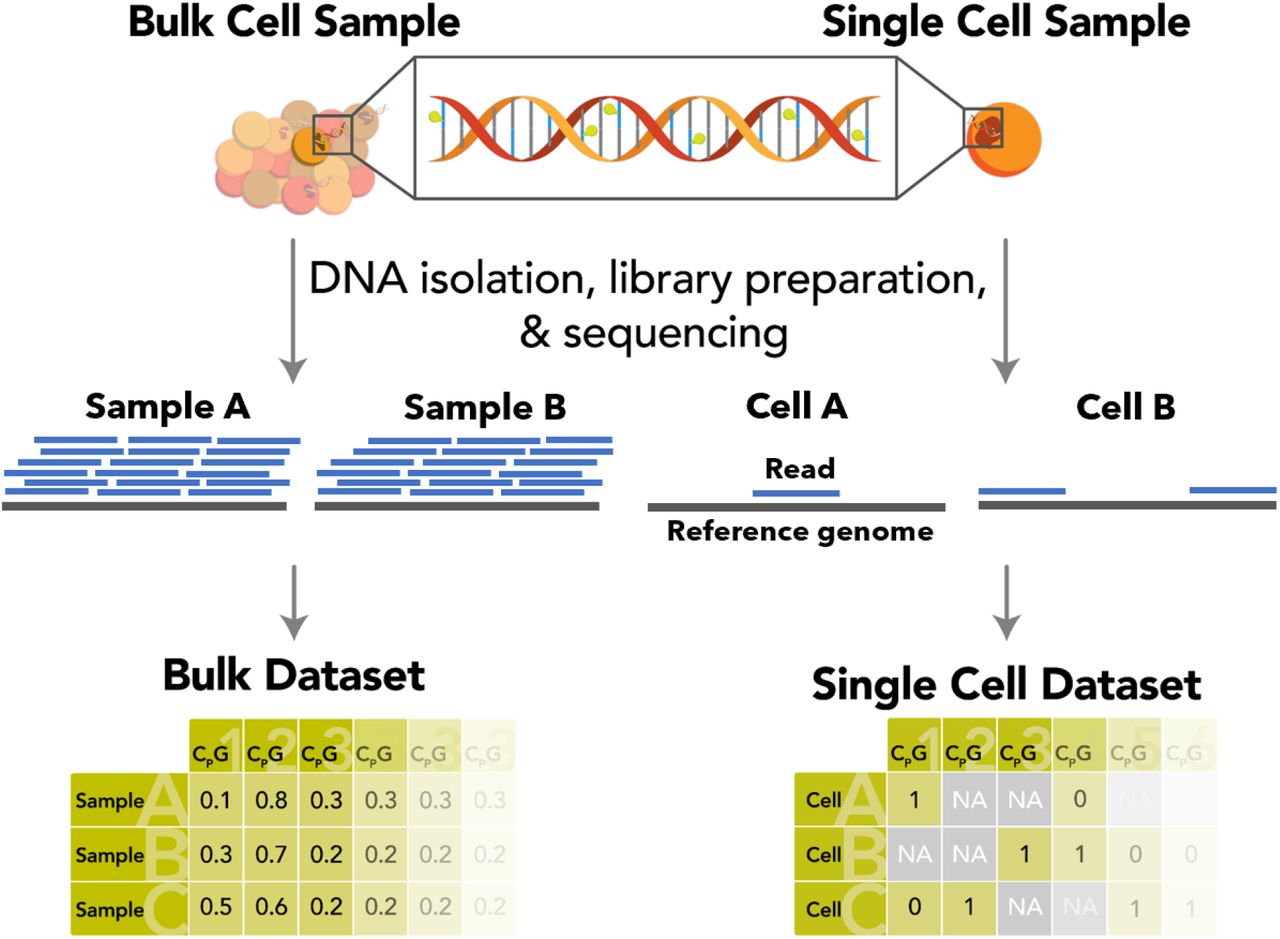
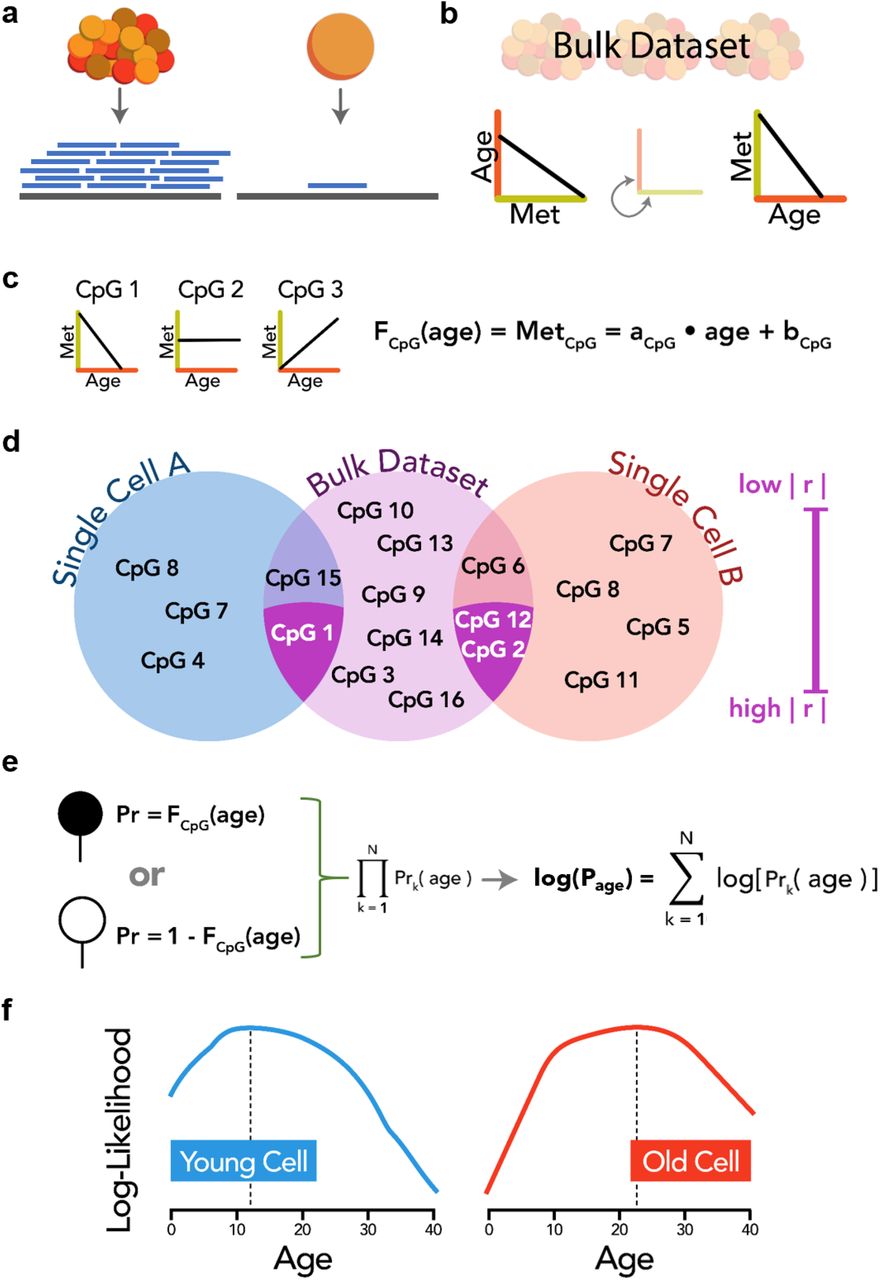
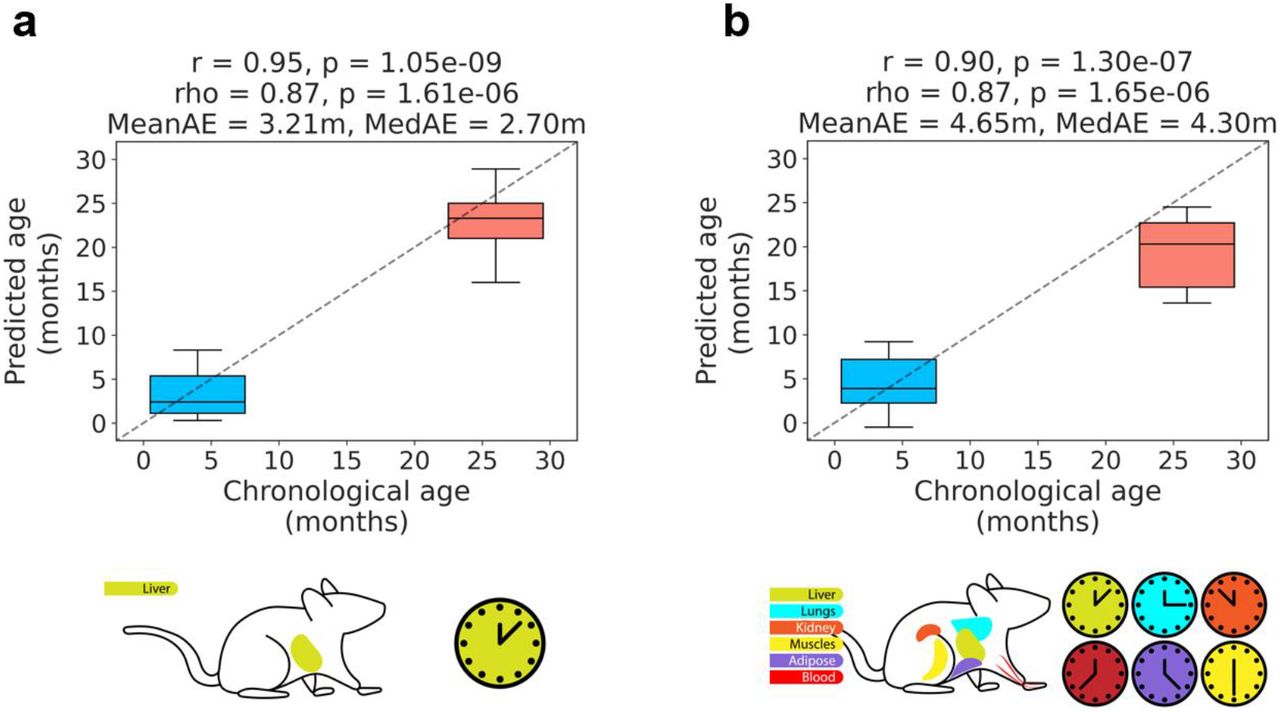
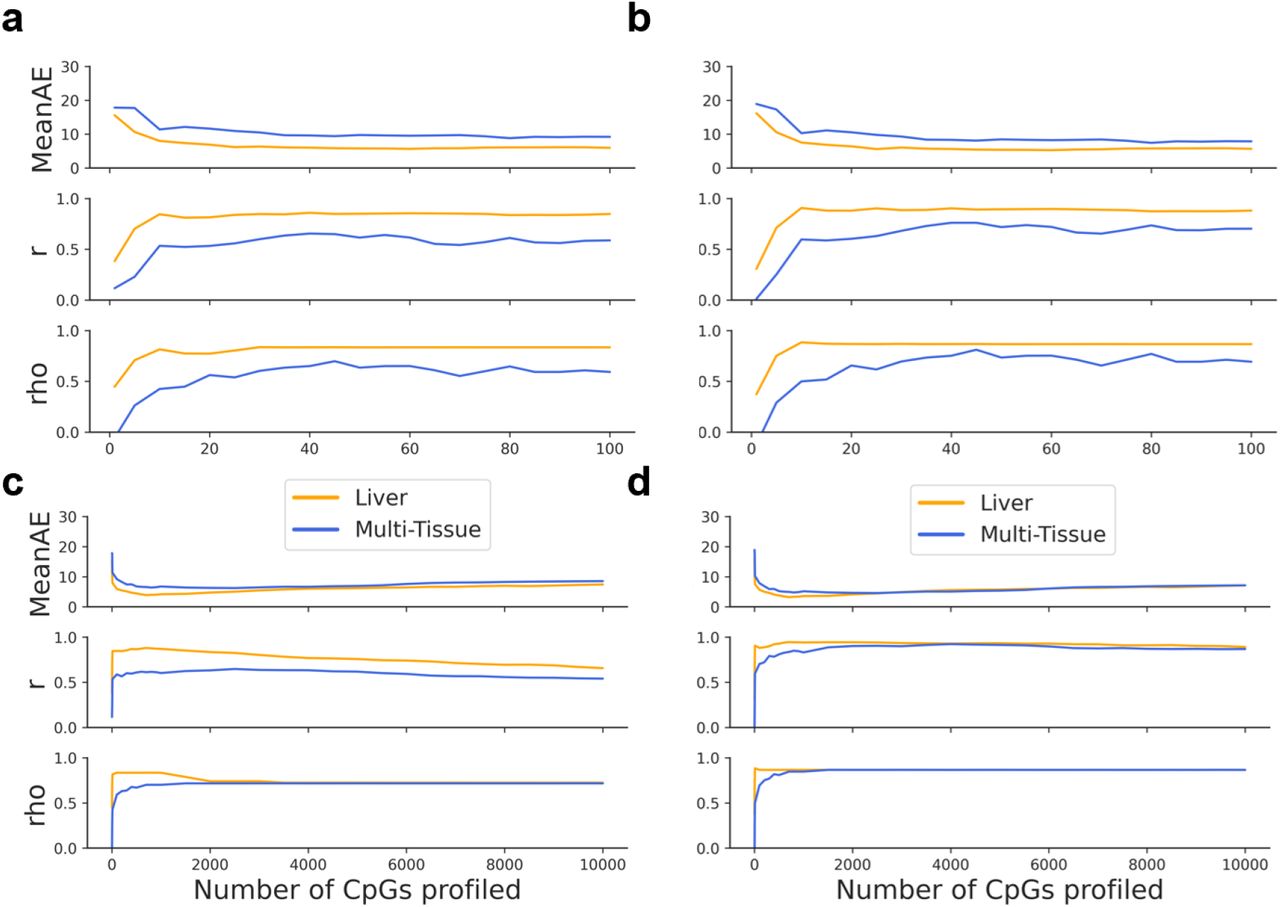
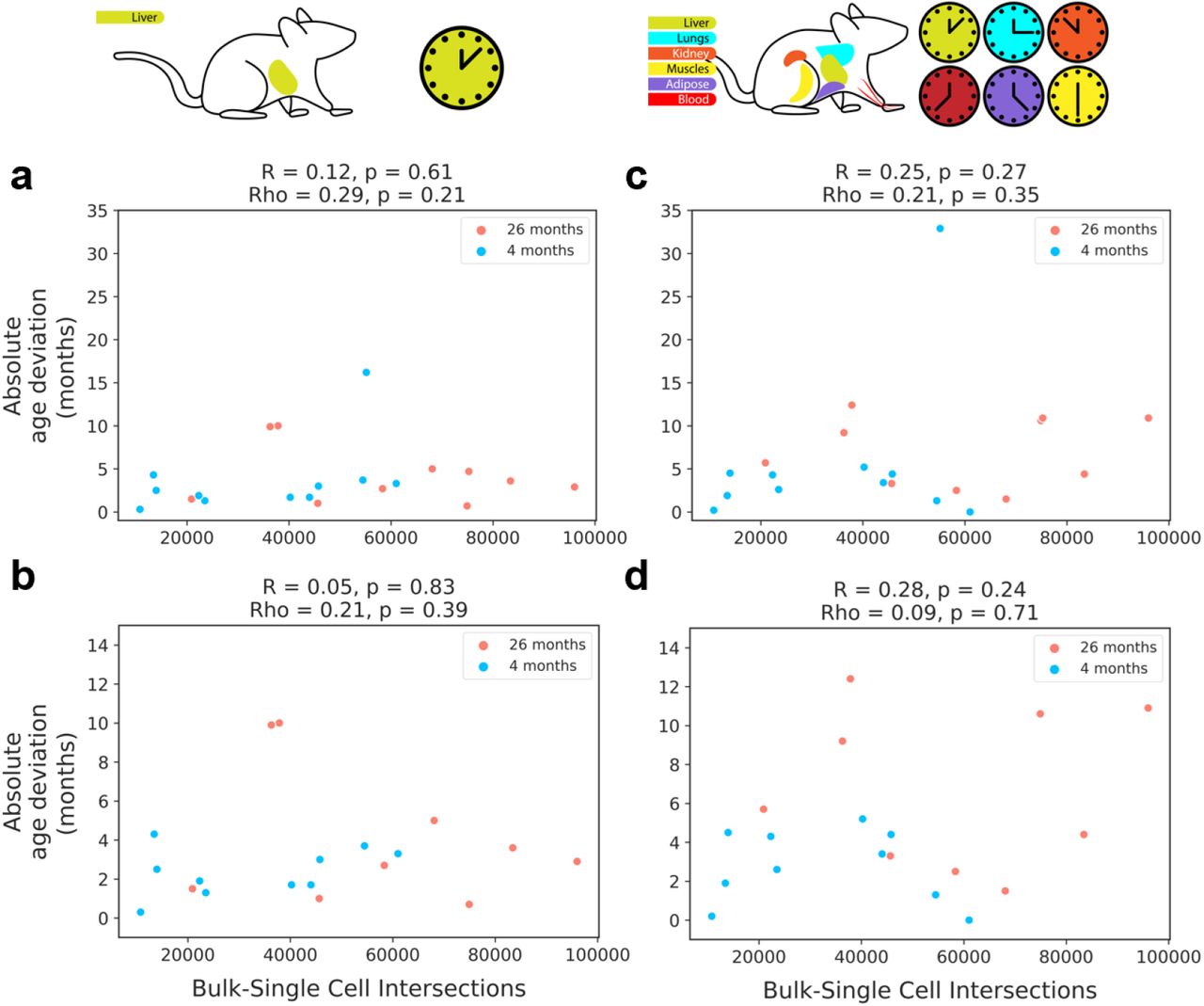
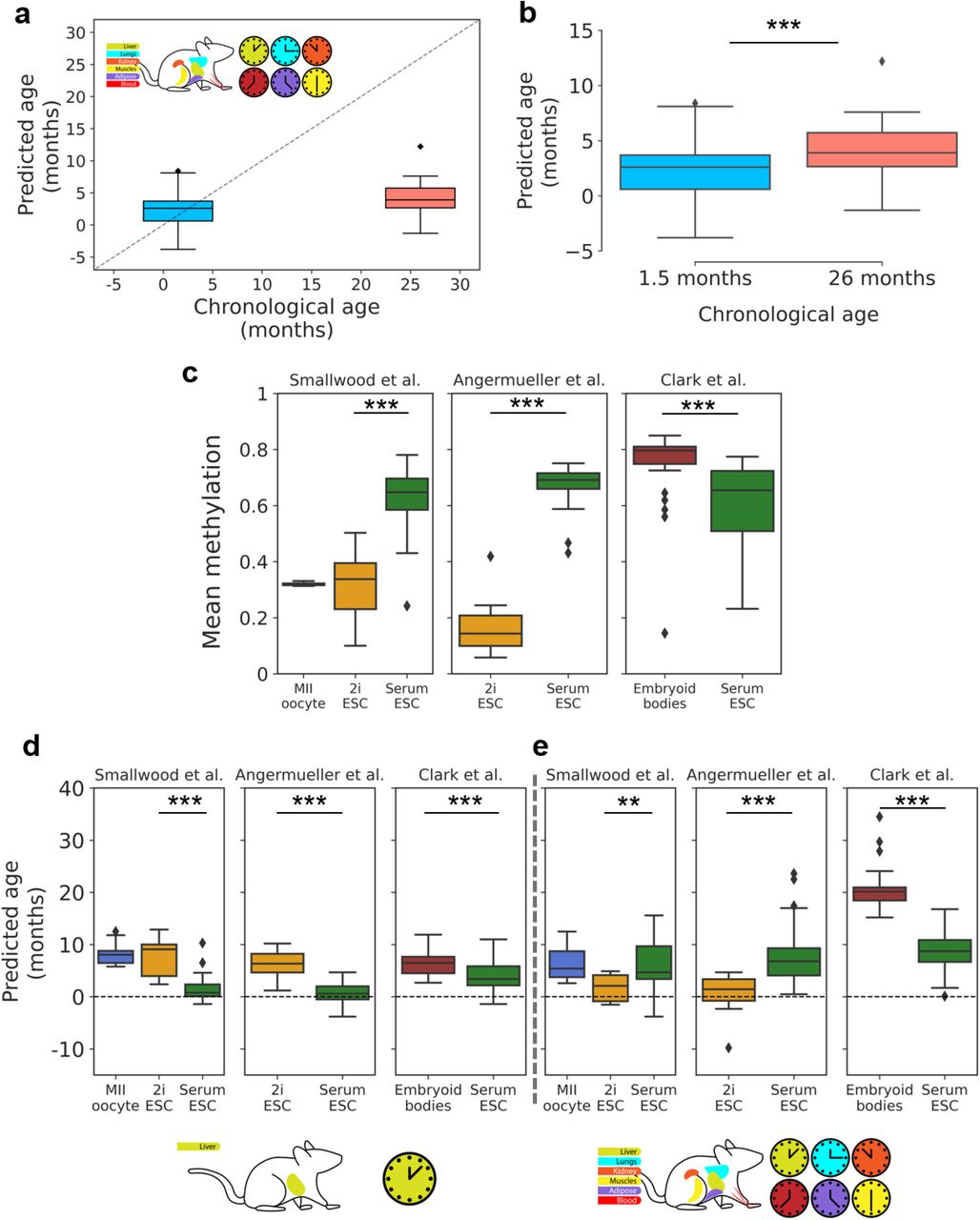
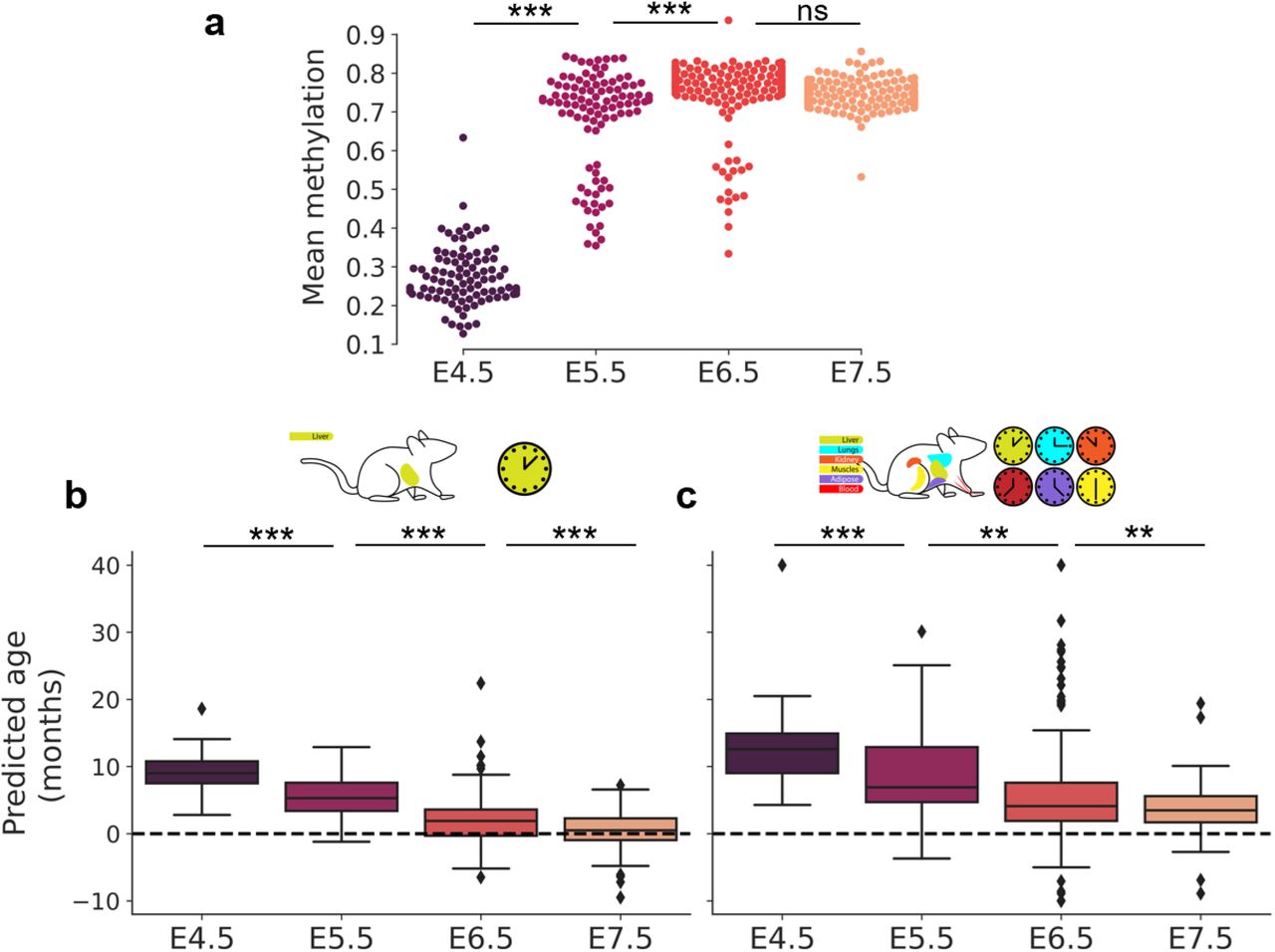
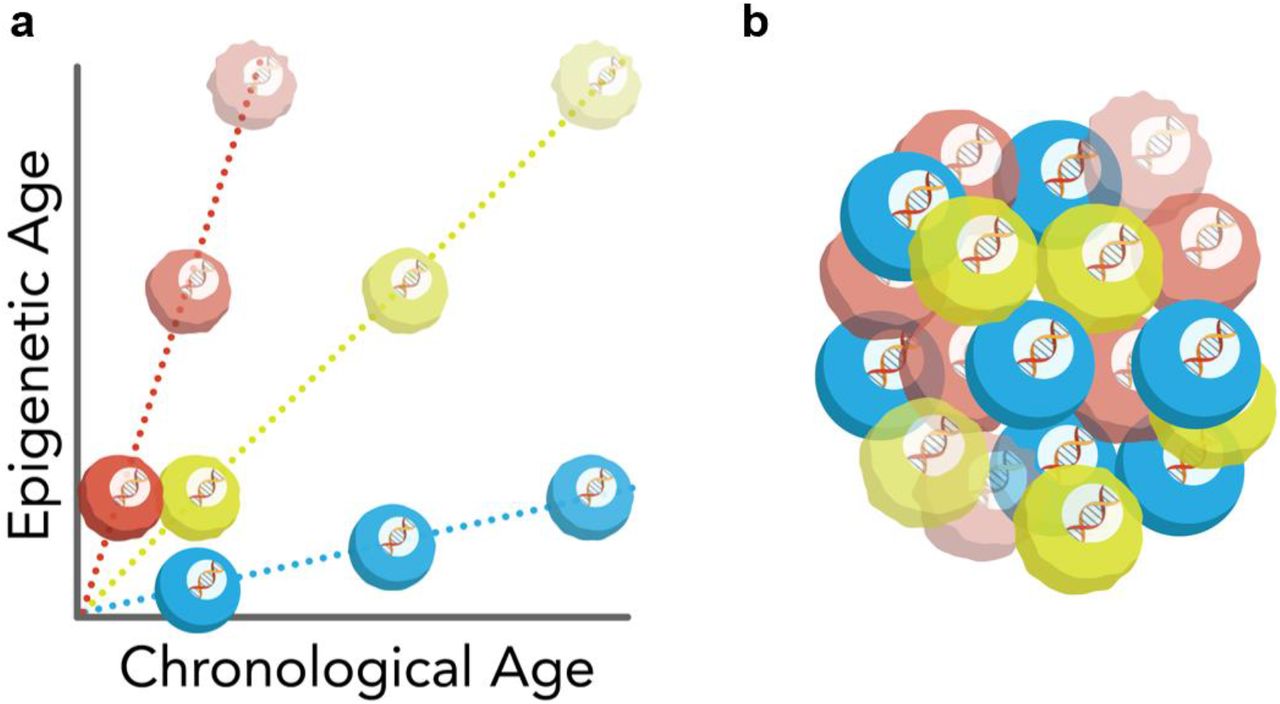
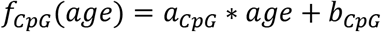 где возраст в месяцах - независимая переменная, f CpG ( возраст ) - прогнозируемый средний уровень метилирования, а a и b - коэффициент линейной регрессии и точка пересечения, соответственно (
где возраст в месяцах - независимая переменная, f CpG ( возраст ) - прогнозируемый средний уровень метилирования, а a и b - коэффициент линейной регрессии и точка пересечения, соответственно ( 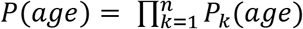 где k представляет собой отдельные CpG. Наша цель - найти максимум этого продукта среди разных возрастов (т.е. найти наиболее вероятный возраст для наблюдения этого конкретного паттерна метилирования). Для этого мы взяли сумму по CpG логарифма вероятностей (чтобы избежать ошибок потери значимости во время вычислений). Это дает нам
где k представляет собой отдельные CpG. Наша цель - найти максимум этого продукта среди разных возрастов (т.е. найти наиболее вероятный возраст для наблюдения этого конкретного паттерна метилирования). Для этого мы взяли сумму по CpG логарифма вероятностей (чтобы избежать ошибок потери значимости во время вычислений). Это дает нам 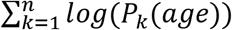 для каждой возрастной ступени. Эти логарифмические суммы обеспечивают показатель вероятности для каждого возраста, когда одна клетка происходит из основной ткани этого возраста. Наконец, мы выбираем возраст максимального правдоподобия в качестве предиктора возраста для одной клетки.
для каждой возрастной ступени. Эти логарифмические суммы обеспечивают показатель вероятности для каждого возраста, когда одна клетка происходит из основной ткани этого возраста. Наконец, мы выбираем возраст максимального правдоподобия в качестве предиктора возраста для одной клетки.